BH12.12/MBGD
提供:TogoWiki
目次 |
RDF化
変更点
- URI内に、Release Date、Ortholog Table ID を組み込む
- <http://.*/orthogroup/¥d+> → <http://.*/2012-01/default/cluster/¥d+>
- オーソログクラスターにOrtholog Table IDをひも付けておいた
- mbgd:clusterOf <http://.*/2012-01/default/cluster>
- オーソログクラスターにOrtholog Table IDをひも付けておいた
- <http://.*/homcluster/¥d+> → <http://.*/2012-01/default/homcluster/¥d+>
- <http://.*/gene/.*/¥d+> → <http://.*/2012-01/default/domain/.*/¥d+>
- <http://.*/orthogroup/¥d+> → <http://.*/2012-01/default/cluster/¥d+>
- その他の変更
- mbgd:from, mbgd:to → mbgd:start, mbgd:end
- mbgd:Orthogroup → mbgd:Cluster
- <http://.*/rdf-schema#> → <http://.*/mbgd.owl#>
- URIのポート番号を除いた
追加データ
- 様々な生物種セットについて、オーソログテーブルを作成
- いくつかのオーソログテーブルを、ロードしてテストした
- tax9(ブフネラ・アフィディコラ, 12 genomes)
- tax809 (クラミジア科, 8 genomes)
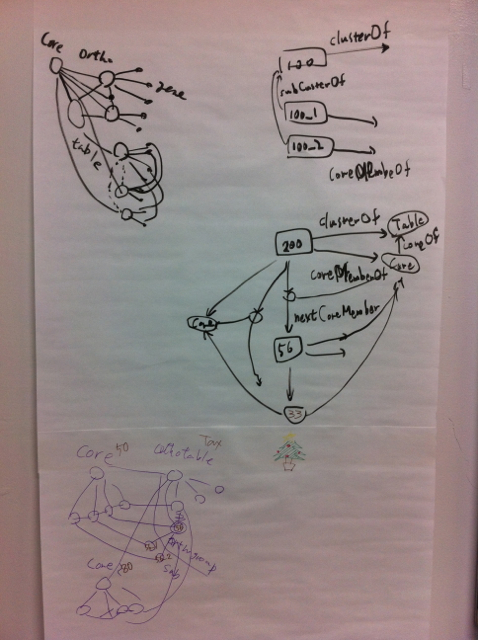
Syntenic Core について
- <SyntenicCore50> :member <Cluster100_1>
- <Cluster100_1> :next <Cluster56>
- <Cluster56> :next <Cluster33>
- <Cluster100> :member <Cluster100_1>
SPARQL
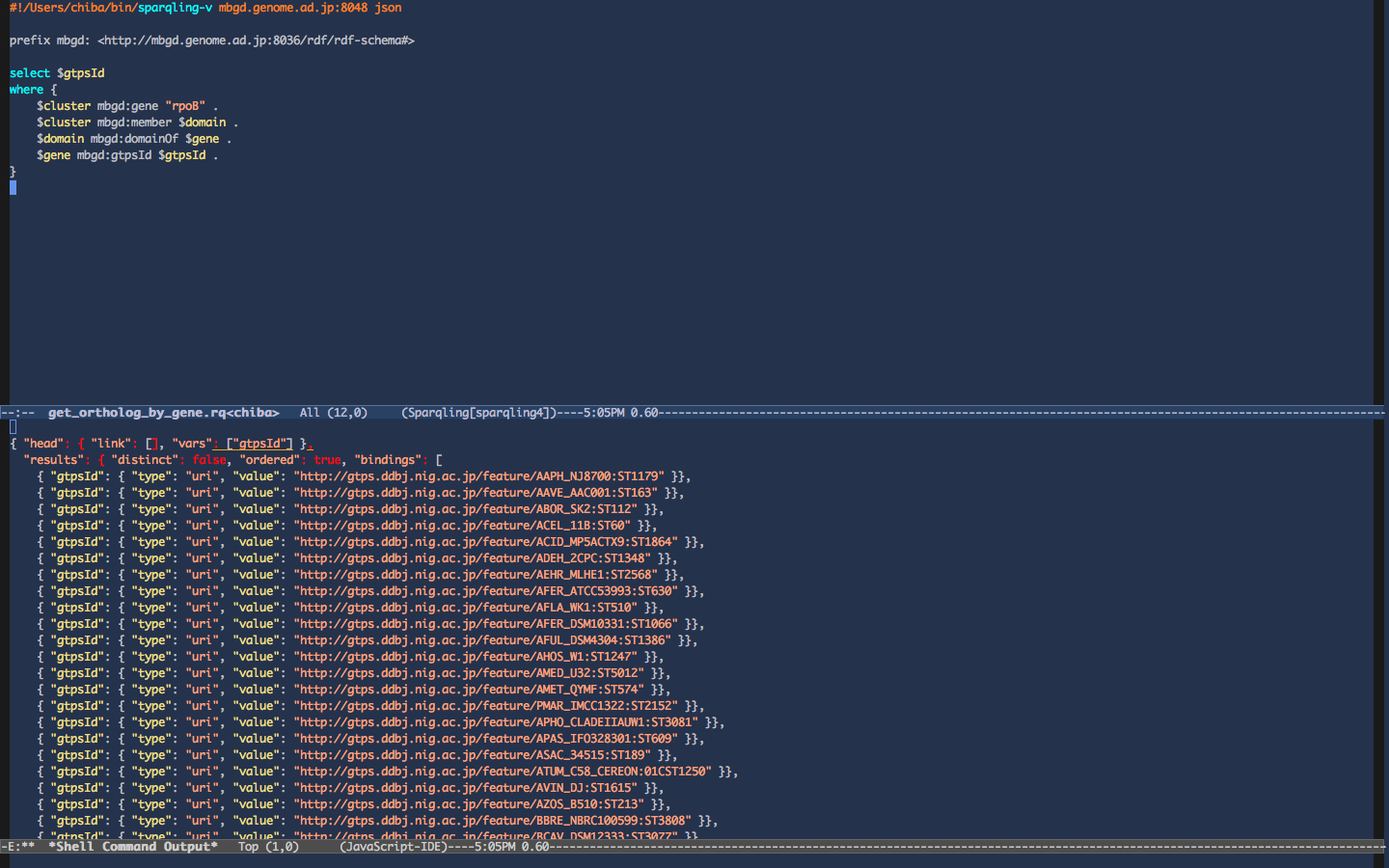
- 特定のオーソログクラスタリング結果を指定して (tax9: クラミジア科, 8 genomes)、rpoBのオーソログメンバーを取得
prefix mbgd: <http://mbgd.genome.ad.jp/rdf/2012-02/mbgd.owl#> select $cluster $gene where { $cluster mbgd:clusterOf <http://mbgd.genome.ad.jp/rdf/2012-02/tax9> . $cluster mbgd:gene "rpoB" . $cluster mbgd:member $domain . $domain mbgd:domainOf $gene . }
- Enterobacteriaceae (Taxonomy ID 543) で保存されているオーソログを知りたい
25 生物種のうち何生物種が持っているかを、オーソログごとに調べ、多い順にソートするSPARQL
define input:inference "http://localhost:8890/DAV/owl/rule_set" PREFIX mbgd: <http://mbgd.genome.ad.jp:8036/rdf/rdf-schema#> PREFIX tax: <http://purl.org/obo/owl/NCBITaxon#> select $cluster count(distinct $genome) as $count where { $cluster mbgd:member $domain. $domain mbgd:domainOf $gene. $gene mbgd:geneOf $genome . $genome a tax:NCBITaxon_543 . } order by desc ($count)
20種(80%)以上が持っている:1,044個のオーソログクラスター
15種(60%)以上が持っている:2,115個のオーソログクラスター
時間:14.7 sec
- 様々なオーソログクラスターのメンバー遺伝子の、アノテーションを取得する
- (注)以下のエンドポイント mbgd.genome.ad.jp:8048 は、Virtuosoのデフォルト設定なので、10,000行までしか出力されない
#!/Users/chiba/bin/sparqling-v mbgd.genome.ad.jp:8048 json prefix mbgd: <http://mbgd.genome.ad.jp:8036/rdf/rdf-schema#> select $cluster $gene $geneDescr where { $cluster mbgd:member $domain . $domain mbgd:domainOf $gene . $gene mbgd:geneDescr $geneDescr . }
http://sparqlbin.com/#0020255c1de383d9e5ecf8f7982c0e73
- メンバー数が10個から20個のクラスターに限定して、遺伝子のアノテーションを取得
#!/Users/chiba/bin/sparqling-v mbgd.genome.ad.jp:8047 json prefix mbgd: <http://mbgd.genome.ad.jp:8036/rdf/rdf-schema#> select $cluster $gene $geneDescr where { $cluster mbgd:member $domain . $domain mbgd:domainOf $gene . $gene mbgd:geneDescr $geneDescr . { select $cluster count(distinct $domain) as $count where { $cluster mbgd:member $domain . } } . Filter(($count >= 10) && ($count <= 20)) } order by $cluster
Emacs sparqling-mode
sparqling-mode.el
(defun sparqling-buffer ()
"Submit the current buffer as a query to a SPARQL HTTP endpoint."
(interactive)
(let (tmp beg end command-line-text mode-text)
(setq tmp (point))
;; check if the first line is shebang line
(if (string= "#!" (buffer-substring-no-properties 1 (min 3 (point-max))))
(progn
;; get the command line
(goto-char (point-min))
(forward-char 2)
(setq beg (point))
(end-of-line)
(setq end (point))
(setq command-line-text (buffer-substring-no-properties beg end))
(backward-word)
(setq beg (point))
(setq mode-text (buffer-substring-no-properties beg end))
;; execute
(save-buffer)
(delete-other-windows)
(shell-command (concat command-line-text " " (buffer-file-name)) nil nil)
(message (concat "Shell Command Executed: " command-line-text " " (buffer-file-name)))
(other-window 1)
(if (string= (buffer-name) "*Shell Command Output*")
(progn
(fundamental-mode)
(if (string= "json" mode-text)
(if (fboundp 'js2-mode)
(js2-mode)
(java-mode)))
(if (string= "xml" mode-text)
(xml-mode))
(if (string= "html" mode-text)
(html-mode))
(setq truncate-lines t)
(other-window 1)
))
))
(goto-char tmp)
))
(define-derived-mode sparqling-mode shell-script-mode "Sparqling"
(define-key sparqling-mode-map [\C-\return] 'sparqling-buffer)
)
(font-lock-add-keywords
'sparqling-mode
'(
("\\?\\(\\w+\\)" . (1 font-lock-variable-name-face nil t))
("\\$\\(ARG\\w+\\)" . (1 font-lock-function-name-face nil t))
("\\$\\(\\w+\\)" . (1 font-lock-variable-name-face nil t))
("select" . font-lock-keyword-face)
("construct" . font-lock-keyword-face)
("where" . font-lock-keyword-face)
("service" . font-lock-keyword-face)
))
Install
- sparqling-mode.ql をEmacs Lispのパスに入れ、下記を.emacsに追加する。
(autoload 'sparqling-mode "sparqling-mode" "Mode for editing SPARQL files" t)
(setq auto-mode-alist
(cons (cons "\\.rq$" 'sparqling-mode) auto-mode-alist))
- 同時に、sparqling-vもインストールしておけば、以下のように、SPARQLの実行までできるようになる。
Usage
- SPARQLファイル(ファイル名が *.rq)を開くと、自動的にsparqling-modeが起動する。
- SPARQLを編集する。自動でindentation、syntax highlightingが行われる。
- SPARQLのキーワードを sparqling-mode.el に登録しておくと、それらの色も変わる。
- (小文字の select, construct, where, service のみ、あらかじめ登録してある。)
- shebangラインに、SPARQLクライアントであるsparqling-vが指定されている場合、以下のようにしてSPARQLの実行までできる。
- SPARQLを編集後 Ctrl + return と叩くと、下に別ウィンドウが開いて、SPARQLクエリの実行結果が表示される。
- shebangラインの末尾にあるフォーマット(json, xml等)に応じて、実行結果のフォーマットは変わる。
- 実行結果のフォーマットに応じてモードが設定される。(json → js2-mode がインストールされていれば js2-mode, なければ java-modeで表示)
- SPARQLを編集後 Ctrl + return と叩くと、下に別ウィンドウが開いて、SPARQLクエリの実行結果が表示される。
Memo
- GWT (Google Web Toolkit) による、Webアプリケーションのユーザインターフェースの実装(ヨハンさん)
- あるファミリーでは、60%以上が持っており、別のあるファミリーでは、20%以下しか持っていない、そういうオーソログは?