TPP-masuya
提供:TogoWiki
| 研究開発課題名 | 生命と環境のフェノーム統合データベース |
| 研究代表者 | 桝屋 啓志 |
| 所属・役職 | 理化学研究所 バイオリソースセンター ユニットリーダー |
| 概 要 | 遺伝子の多様性の結果として現れる生命の表現型(フェノタイプ)の情報を、モデル動物(マウス、ラット、ゼブラフィッシュ、メダカ)、ゲノム編集研究など、幅広い研究コミュニティから収集し、研究分野の垣根を超えて標準化・統合化・体系化してオープンに公開する。集約されたフェノタイプ情報は、ゲノム情報や分子情報とともに横断的に利活用することで、新たな生命科学イノベーションの原動力となると期待される。 |
目次 |
参加メンバー
- 桝屋 啓志 (理研)
- 高月 照江(理研)
- 熊谷 禎洋(日立ソリューション)
検討課題
フェノームのRDFスキーマの設計、および相互運用性の確立
- 数値のRDF記述についての検討
- 生物種、系統のRDF(with 植物グループ)
SPARQLthon21#Taxonomy Ontology について
- その他植物グループとのスキーマ検討
- 複数生物のフェノームを繋ぐデータ
- データ項目のリスト
- 外部RDFを利用するデータリスト
TPP_Masuya RDFデータ/スキーマの説明(テスト作成)
(プロパティは、全てを列挙できている訳ではないので注意)
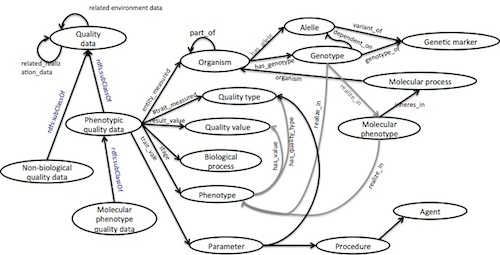
- 表現型データ
計測データオブジェクト毎にインスタンスを作成する。各データは何かのクラスに属するようにする(例:BRCマウスクリニックデータ、等)。計測対象が同じでも、値の形式が異なる場合(定量値と定性値等)は、別のインスタンスを作成する(<=要検討)。本「データ」は、表現型そのものではなく、表現型を[EAVモデル] の形式で記述した「informational entity」である。下記のプロパティを持つ。各プロパティは、なるべくインスタンス生成なしに、クラスで記述できる事を意図してデザインしている。(必要な場合には、プログラム的に生成可能なはず・・)
- indicates_entity_whole プロパティ:測定対象の個体/コホートのインスタンスを述語とする。indicates_entityの解剖学概念やプロセス、indicates_traitのPATOのインスタンスを作らないようにする意図でこのプロパティを使う。Strainそのものを述語にしてはダメ。
- indicates_entity プロパティ:上記プロパティより詳細に、測定対象部分やプロセスを述語とする。述語はクラスで良い。例えば、"物"であるMA, FMA概念や、プロセスであるGOの概念。
- indicates_trait プロパティ:測定対象のQuality Typeを述語とする。述語はクラスで良い。具体的には、PATOの中間概念(attribute slim)。indicates_entityが"物"の場合は、PATO:0001241(physical object quality)、プロセスの場合は、PATO:0001236(process quality)の下位概念とすること。遺伝子発現の場合は、組織における転写産物の#濃度、と考えるので、physical object qualityである。
- indicates_value プロパティ:測定結果であるQuality Valueを述語とする。述語はクラスで良い。定性値の場合には、PATOの最下位の概念(value silm)または、MPやMPOの最下位の概念。定量値の場合には、TPP-DB#数値/定量値の通りに書く。
- indicates_stage プロパティ:測定された発生ステージを述語とする。述語はクラスで良い。
- そのデータが引き起こされる原因となりそうなデータをつなぐプロパティ:名称検討中。述語は表現型データ
- envirionmentプロパティ:環境データを述語とする。環境データもEAVで書きたい。
- その他skosの著者や測定日等を示すプロパティを用いても良い。
- バイオリソース/サンプル/Biological Entity
- Biological Strain:TPP-DB#biological entityの通りに書く。寄託者など、バイオリソース特有のプロパティもつける。
- taxonプロパティ:TPP-DB#biological entityの通り。
- has_alleleプロパティ:述語が#アレル、変異遺伝子。
- depositorプロパティ(バイオリソース特有):述語は、寄託者あるいは寄託施設のインスタンス。
- BRC_IDプロパティ等(バイオリソース特有:述語はリテラル。他の括りのIDも同様にリテラルプロパティで作る。
- コホート:コホートを定義し、インスタンスを作成する。系統や性別などコホートの定義に関わるプロパティをつける。
- memberプロパティ:系統を述語とする。コホートのメンバーのクラス制約が、系統クラスである、という意味。(OWLのプロパティにした方が良い?)
- grouping_genderプロパティ:基本的に、PATO:0001894(phenotypic sex)の下位概念を述語とする。
- has_genotypeプロパティ:述語が#genotype。グルーピングの基準がgenotypeである場合。
- 名称、別名関係:skos:preflabel, rdfs:label, skos:altlabelを使用する。
- Biological Strain:TPP-DB#biological entityの通りに書く。寄託者など、バイオリソース特有のプロパティもつける。
- 個体:ブランクノードとする。「ディッシュの中の細胞」についてもコホート様のものを定義するのもどうかと思われ、ブランクノードで良いのではないか・・・
- 濃度:基本的に、PATO:0000033(concentration of)と考える。遺伝子発現もこれ。
- concentration_ofプロパティ:(かぶってておかしいけど他に名前が思いつかないので・・)述語はmolecular entity。ChEBI、 TPP-DB#化合物など。
- allele:アレル
- variant_ofプロパティ:述語が遺伝子あるいは、ゲノム領域
- genotype:遺伝子型/遺伝型。表現型と同様、性質と定義する。具体的なタイプを示すので、性質値である。
- dependent_ofプロパティ:性質が依存するモノへのリンク。genotypeの場合、alleleが述語。2倍体であれば、dependent_ofが2つとなる
- 文献へのリンク:TPP-DB#文献
- 種間表現型のリンク:検討中。PhenomeNet, Uberon, Uberpheno, Monarch Initiative, Aber-OWLのデータを駆使したい。
- 疾患
- 画像
画像を主語としたRDFを作る。プロパティはfoafのものを使う。
- foaf:depictsプロパティ:画像から画像に写っているもの、主題等へのリンク。
これまでの成果
- フェノームのRDFスキーマの設計:全体概要像(2014.6.19更新)
- フェノームのRDFスキーマの設計:基礎部分: EAVモデル(2014.6.19更新)
- 数値のRDF記述についての検討(2014.8.18更新)
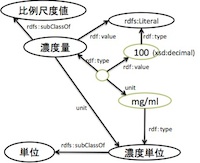
- 方法1:ブランクノードを使った記述
[ a:濃度量; :value “100”^^xsd:demical; :unit <mg/ml> ]こんな感じ?
- 黒川Gも同じ方法。神崎氏の教科書 [http://milicicvuk.com/blog/2011/08/16/literals-blank-nodes-n-ary-relations-and-rdfvalue/ 他の記事」
- 利点:100mg/mlを誰もが勝手に作っても問題無い。100mg/mlを比例尺度として、mg/mlを単位としてそれぞれオントロジーで管理できる。
- >>mg/mlという単位自体を、比例尺度とする考え方もあるのではないか。
- 不利な点:プロパティが2つ多い。
- 方法2:^^を使ってリテラルに属性を書く
- W3CのSPARQLの記事 に紹介されている(2.3 Matching RDF Literalsのところ)
- 方法2:^^を使ってリテラルに属性を書く
:has_quantity “100”^^xsd:demical; ^^mg/ml;
(has_quantityはリテラルプロパティ) こうすることで、リテラルでありながら、mg/mlである、という意味をもつことができる。
- 利点:プロパティが少なくて済む
疑問点:xsd:demicalはリテラルの下位概念で良いと思うが、mg/mlはリテラルとは全く違う概念定義されるはずなので、それは良いのかどうか
- 生物種、系統の記述についての検討(with 植物グループ)(2014.8.18更新)
- データベースリスト で検討したものに従う。
- データ項目のリスト
植物グループに習って作成する。
●登録を予定している項目のリスト(2014.7.16更新:高月)
https://docs.google.com/spreadsheets/d/161D5D1Vp00QJs9AaaHXJULAiqapL3xkibKmltmroObQ/edit#gid=sharing
- 外部RDFを利用するデータリスト(2014.7.16更新:高月)
外部からのRDFを利用する場合、直接のリンクは、動作に不具合が生じることがあるとの事。 Bioportalでは、エンドポイントを持っているが、基本的にはデモ用のメンテナンスとの事で、 大量のバッチを投げたりすることは推奨しておらず。 また、Bio2RDFのサイトでは、エンドポイントも設置してあるが、保持しているデータが古く 更新されていない。
●現在までの調査結果(2014.7.16更新:高月) https://docs.google.com/spreadsheets/d/1pl6szC4lK9qJzXxMbFwg1DrvPPRJ85NoygzcOyudmwo/edit?usp=sharing
●バイオリソースを記述するための各種オントロジーについて(2014.10.28)
*1:biorel http://terms.tdwg.org/wiki/Biorelations (上位概念として設定?)
- 実験系を記述するためのオントロジー(RGDでの開発)
*2:xco http://bioportal.bioontology.org/ontologies/XCO *3:MMO http://bioportal.bioontology.org/ontologies/MMO *4:CMO http://bioportal.bioontology.org/ontologies/CMO
*5:EFO http://www.ebi.ac.uk/efo/
*6:ECO http://www.ebi.ac.uk/ontology-lookup/browse.do?ontName=ECO
(アノテーションの評価に利用できそう) 実際にORDOで利用されている