SPARQLthon22
提供:TogoWiki
第22回 SPARQLthon を下記日程で開催したいと思います。
目次 |
開催概要
- 開催期間:2014年7月15日(火) 10:00 〜 16日(水) 18:00
- 開催場所:ライフサイエンス統合データベースセンター (DBCLS) @ 東京大学 柏の葉キャンパス駅前 サテライト 6階
- アクセス:http://dbcls.rois.ac.jp/access
- 開催連絡:http://groups.google.com/group/biohackathon-jp メーリングリストにて
プロジェクト
TPP グループ全体
各データベースの該当分野とモデル図
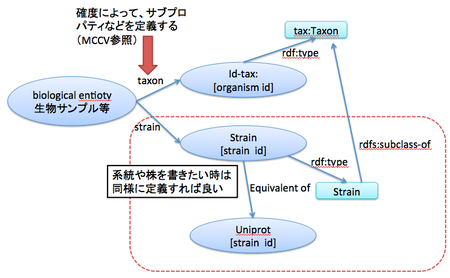
- taxonomy ontologyの階層を利用したい
- <organism> :taxon <http://identifiers.org/taxonomy/【taxid】>
- :taxon については要検討、ゲノムRDFの場合、insdc:db_xref、rdfs:seeAlsoを利用している
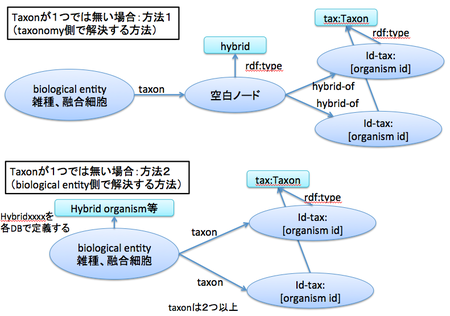
- ハイブリット植物、融合細胞、微生物ープロファージ、雑種などhypbridな生物種を表現したい
- :taxon (:hybrid-of) なpredicateを用いて2つ以上のを <http://identifiers.org/taxonomy/【taxid】> をリンクする
- RDFモデル2つの案、Taxonomy側で解決する or biological entity側で解決する
- tax-idがなくてもリンクしたい
- 上位ランクのtaxidをアサインする
- MCCVで記述しているようなtaxidのアサインの確度をpredicateで分ける、skos:mappingRelationのsubproperty(skos:closeMatch, skos:exactMatch, skos:broadMatch, skos:narrowMatch, skos:relatedMatch)のようなイメージ
- SPARQL問い合せのために、定義したsubpropertyも冗長にトリプルをもつ必要があるかもしれない
- Strain相当のクラスを独自に定義したとき、NCBIやUniProtのTaxonomyオントロジーの Taxonomic Rankのクラスとの関係を記述したい
- :equivalent-of で記述?
- 川島さんの意見:(2014/8/18追加)hybridな場合と、そうでない場合で、スキームが共通なほうが、SPARQLが書きやすいのではないか(イレギュラー対応が少ない)
- taxon のサブクラスとしてのhybridとは何か、という定義の問題も出てくる。
- hybrid生物がtaxonプロパティを2つ持つ方が、素直で(hybridでない場合と一致していて)SPARQLを書きやすい/使い回しやすいのでは?
構造の明確な場合(InChIなどで表現できる)と不明確な場合(Mwのみ、分類まで)を分けて考える。
- Chemical Informaiton Ontology を利用 (櫛田・山田)
- PubChem(NCBI) , ChEMBLでも利用されている。
- InChI & InChIKey
@prefix cheminf: <http://semanticscience.org/resource/> .
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
@prefix db: <http://testdb.jp/compound/>
<db/0001#standard_inchi> a cheminf:CHEMINF_000113 ;
cheminf:SIO_000300 "InChI=1S/C6H12O6/c7-1-2-3(8)4(9)5(10)6(11)12-2/h2-11H,1H2/t2-,3-,4+,5-,6-/m1/s1" ;
rdfs:label "0001 Standard InChi" .
<db/0001#inchikey> a cheminf:CHEMINF_000059 ;
cheminf:SIO_000300 "inchikey/WQZGKKKJIJFFOK-VFUOTHLCSA-N" ;
rdfs:label "0001 inchikey" .
- Molecular Weight
- CHEMINF:000350 molecular weight of the corresponding free base
- CHEMINF:000216 average molecular weight descriptor
- CHEMINF:000217 exact mass descriptor
- CHEMINF:000218 monoisotopic mass descriptor
@prefix cheminf: <http://semanticscience.org/resource/> .
<http://rdf.ebi.ac.uk/resource/chembl/molecule/CHEMBL6939#mw_freebase> a cheminf:CHEMINF_000350 ;
cheminf:SIO_000300 "293.4"^^xsd:double .
- CAS Registry Number(CASとのライセンスに注意してください)
@prefix cheminf: <http://semanticscience.org/resource/> .
@prefix db: <http://db.jp/compound/> .
<db/0002> a cheminf:CHEMINF_000446 ;
cheminf:SIO_000300 "cas_rn_001" .
- 日化辞をハブとして活用したい
- Dictionary of Natural Products の項目を利用
- DNPの項目をChEBI ontologyを調べて関連付ける作業を実施(時松・山田)
TPP グループ
- 生物種メタボロームモデル・データベースの構築 (有田)
- MassBank サーバを遺伝研に移転 (有田)
- KNApSAcK における生物種の情報 (金谷)
- 糖脂質の ID を付与 -- 740 件
- ゲノム・メタゲノム情報統合による微生物DBの超高度化推進 (黒川)
- バクテリアゲノムのデータ追加 -- PDO (感染症) を改訂 (山本)
- 疾患ヒトゲノム変異の生物学的機能注釈を目指した多階層オミクスデータの統合 (菅野)
- 健常人やがん細胞株などの ChIP データ、DBTSS データの RDF 化 (富所)
- 植物ゲノム情報活用のための統合研究基盤の構築 (田畑)
- スキーマ設計 -- パワーポイントで図を生成したので検討 (平川・市原)
- 糖鎖統合データベースおよび国際糖鎖構造リポジトリの開発 (成松)
- 桝屋シートをもとに RDF データで連携できる点を検討 (全員)
- TPP と化合物の RDF 化について相談 (山田)
- 糖鎖リポジトリ開発(青木・新町)
- 生命と環境のフェノーム統合データベース (桝屋)
- 疾患と文献 (桝屋)
- 複数の phenome を連携するための既存の公共データを調査 (熊谷)
- データ項目と外部の RDF データで利用可能なものを調査 (高月)
SPARQLthon グループ
- オーソログの RDF を対象にした SPARQL 開発 (千葉)
- SPARQL ビルダの論文投稿 (小林)
- がんゲノムの RDF 調査 (山中)
- ゲノム RDF のメタデータ調査 -- NCBI の Assembly reports に BioProject <-> Accession ID の対応 (藤沢)
- DDBJ と NBRC のバイオサンプル一括登録のための属性情報、菌株 (藤沢・川島)
- TogoGenome 更新、TogoStanza 開発 (守屋・片山)
- 反応オントロジーの確認 (時松・櫻井)
- スキーマ図の方針を考える (大田)
- 医療安全の OWL, 医療知識, 患者横断的な検索 (河添)
- TripleDataProfiler更新や関連ツール調査([1],[2],[3])など (山本)
参考リンク
参加者
- 片山俊明(DBCLS)
- 川島秀一(DBCLS)
- 小林紀郎(理研)
- 永野朗夫(PENQE)15日のみ
- 藤澤貴智(遺伝研)
- 岡別府陽子(MSS)
- 河添悦昌(東大病院)
-
中村保一(遺伝研)16日のみすみません、急な打合につき出席撤回します --yn - 千葉啓和(基生研)15日のみ
- 戀津魁(理研)
- 守屋勇樹(DBCLS)
- 高月照江(理研 BRC)
- 櫛田達矢(NBDC)15日のみ
- 櫻井望(かずさ)15日のみ
- 桝屋啓志(理研)15日のみ
- 今井 健(東大病院)15日のみ
- 時松敏明(DBCLS)
- 山田一作(野口研)
- 山口敦子(DBCLS)
- 大田達郎 (DBCLS)
- 山本泰智 (DBCLS)
- 平川英樹(かずさ)15日のみ
- 市原寿子(かずさ)
- 山本 希(東工大)15日のみ
- 木下聖子(創価大)15日のみ
- 青木信行(創価大)15日のみ
- 新町大輔(創価大)15日のみ
- 山中遼太(先端研)