SPARQLthon35
提供:TogoWiki
第35回 SPARQLthon を下記日程で開催したいと思います。
目次 |
開催概要
- 開催期間:2015年 8月10日(月) 10:00 〜 11日(火) 18:00
- 開催場所:遺伝学研究所 生命情報研究センター棟 (DDBJ棟) 4F会議室 (W403-405) + 生命情報研究棟西棟 DBCLS
- アクセス:http://dbcls.rois.ac.jp/access
- 遺伝研行きのシャトルバスがあります。乗り口は北口から出てロータリー向かって左手です。
- 新幹線ーNIGシャトルバス接続情報
- 9:30出発のシャトルバスに乗ってお越しいただくと、DDBJ棟までご案内します。
- 関東方面からお越しの方は こだま639号 (東京8:26発、品川8:34発) がスムーズです。
- 関西方面からお越しの方は こだま632号 (名古屋7:29発) がスムーズですが、朝が早いので前泊する方が楽です。
- お昼について
- 所内には小さい食堂がありますが、売店などはありません。お弁当などをお持ち頂くことをお勧めします。
- 参考: ランチスポット情報 [三島グルメ]
- 所内には小さい食堂がありますが、売店などはありません。お弁当などをお持ち頂くことをお勧めします。
- 夕食について
- 初日の夕食は参加者のみなさまで三島駅前に出かける予定です。
- ホテルについて
- 駅の周りのホテル
- ホテルアルファワン http://www.alpha-1.co.jp/mishima/ 南口。INSDCミーティングの時に使ってるホテルその1
- ホテルマッシモ三島 http://www.massimo-m.jp 南口。INSDCミーティングの時に使ってるホテルその2
- 東横イン http://www.toyoko-inn.com/hotel/00215/ 北口。あんまりお店がない方だけど遺伝研行きシャトルバス乗り場に近い方
- ホテルブルーム http://www.jalan.net/yad314620/ 南口。ちょっと怪しげだが安い
- ホテルセレクトイン http://select-hotels.jp/pc/hotel/mishima/ 南口というよりは三嶋大社近く
- 照明館 http://www.shoumeikan.com/ 南口。繁華街の中
- みしまプラザホテル http://www.mishimaph.co.jp/ 一番高級
- ドーミーイン http://www.hotespa.net/hotels/mishima/ 南口。温泉があるらしい。
- 駅の周りのホテル
- 開催連絡:http://groups.google.com/group/biohackathon-jp メーリングリストにて
プロジェクト
今日の課題は → http://tinyurl.com/sparqlthon35nig へ、進捗はこちらの Wiki に(必要なら SPARQLthon35/サブページ のリンクを作って)ご記入ください。
TPP グループ全体
- 糖鎖統合データベースおよび国際糖鎖構造リポジトリの開発 (成松)
- 糖鎖のオントロジーであるGlycoRDFをRDFizingDatabaseGuideline へ記載(山田)
- "バージョン 1.3 as of "2015-08-10"^^xsd:date"とした。 (生命科学情報に関するドメインオントロジーにGlycoRDFを追加したので1.2 -> 1.3 とした)
- 糖鎖のオントロジーであるGlycoRDFをRDFizingDatabaseGuideline へ記載(山田)
- GlyTouCanでMS値の表示、計算に問題があったので molecular mass descriptor(SIO)について相談(山田・有田・吉沢)
- MSデータと比較することできるように monoisotopic mass descriptor をGlyTouCanでも利用する予定: Glycan詳細画面のmass -> monoisotopic massへの変更
- 参考:
- Orbitrap質量分析(pdf) が高分解能
- http://webs2.kazusa.or.jp/mfsearcher/atomlist
- http://semanticscience.org/resource/CHEMINF_000218 <- monoisotopic mass descriptor
- 参考:
- MSデータと比較することできるように monoisotopic mass descriptor をGlyTouCanでも利用する予定: Glycan詳細画面のmass -> monoisotopic massへの変更
- GlyTouCanでMS値の表示、計算に問題があったので molecular mass descriptor(SIO)について相談(山田・有田・吉沢)
- 化合物構造のRDF化、SIO調査(山田・有田)
- 化合物構造に利用できそうな部分構造
- has component part [SIO_000369]
- has proper part [SIO:000053]
- 部分構造の結合に利用できそうなもの
- is connected to [SIO:000203] > is directly connected to [SIO:000652] > is covalently connected to [SIO_000334]
- SIO:000203: A is connected to B iff there exists a fiat, material or temporal path between A and B.
- SIO:000652: A is directly connected to B iff there exists a path direclty between A and B.
- SIO_000334: is covalently connected to is a relation between an atom and another atom.
- 化合物構造に利用できそうな部分構造
- 化合物構造のRDF化、SIO調査(山田・有田)
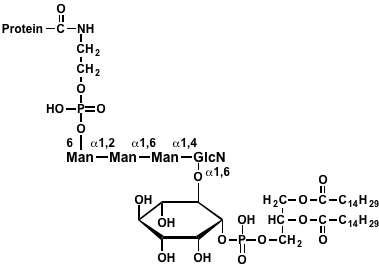
- GPIアンカーのRDF化の検討
@prefix sio: <http://semanticscience.org/resource/> .
@prefix faldo: <http://biohackathon.org/resource/faldo> .
@prefix uniprot: <http://purl.uniprot.org/core/> .
@prefix glycan: <http://purl.jp/bio/12/glyco/glycan#> .
# has component part [SIO_000369]
<http://np.org/GPIanchor>
sio:SIO_000369 # has_component_part
<http://natpros.org/peptide>
, <http://natpros.org/sm/OP^XOCCN/2O/2=O>
, <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1>
, <http://np.org/cyclitol_residue/inositol(1P_hxh_2C14_3C14)>
, <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1> .
# is component part of [SIO_000313]
<http://natpros.org/peptide>
sio:SIO_000313 # is_component_part_of
<http://np.org/GPIanchor> .
<http://natpros.org/sm/OP^XOCCN/2O/2=O>
sio:SIO_000313 # is_component_part_of
<http://np.org/GPIanchor> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1>
sio:SIO_000313 # is_component_part_of
<http://np.org/GPIanchor> .
<http://np.org/cyclitol_residue/inositol(1P_hxh_2C14_3C14)>
sio:SIO_000313 # is_component_part_of
<http://np.org/GPIanchor> .
<http://np.org/cyclitol_residue/inositol(1P_hxh_2C14_3C14)>
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSaloc> .
<http://natpros.org/sm/OP^XOCCN/2O/2=O>
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSdloc> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1>
sio:SIO_000313 # is_component_part_of
<http://np.org/GPIanchor> .
<http://natpros.org/peptide>
glycan:has_AA_sequence "QPFNVTQGKYSTAEECSADSDLNFLIPVAVG" ;
# glycan:has_glycosylated_AA <http://natpros.org/peptide#N4> .
sio:SIO_010074 # amino_acid_residue
<http://natpros.org/peptide#N4> .
<http://natpros.org/peptide#N4>
a sio:SIO_010074 ; # amino_acid_residue
# glycan:Glycosylated_Amino_Acid <http://natpros.org/peptide#N4> ;
faldo:location <http://natpros.org/peptide#N4loc> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1>
glycan:has_glyco_sequence "WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1" ;
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSa> ;
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSd> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSa>
# a sio:SIO_010334 ; # carbohydrate residue
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSaloc> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSd>
# a sio:SIO_010334 ; # carbohydrate residue
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSdloc> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSaloc>
faldo:position 1 ;
rdf:type faldo:ExactPosition .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1#MSdloc>
faldo:position 4 ;
rdf:type faldo:ExactPosition .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1>
# glycan:has_glyco_sequence "WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1”;
faldo:location <http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1#MSa> .
# is_directly_connected_to [SIO:000652]
<http://natpros.org/peptide>
sio:SIO_000652 # is_directly_connected_to
<http://natpros.org/sm/OP^XOCCN/2O/2=O> .
<http://natpros.org/sm/OP^XOCCN/2O/2=O>
sio:SIO_000652 # is_directly_connected_to
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/2,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5]/1-2-2-2/a4-b1_b6-c1_c2-d1>
sio:SIO_000652 # is_directly_connected_to
<http://np.org/cyclitol_residue/inositol(1P_hxh_2C14_3C14)> .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1>
sio:SIO_000652 # is_directly_connected_to
<http://np.org/cyclitol_residue/inositol(1P_hxh_2C14_3C14)> .
#
<http://natpros.org/sm/OP^XOCCN/2O/2=O>
faldo:location <http://natpros.org/peptide#N4loc> .
<http://natpros.org/peptide#N4loc>
faldo:position 4 ;
rdf:type faldo:ExactPosition ;
faldo:reference <http://np.org/seq> .
# <http://np.org/seq> a uniprot:Sequence ;
# rdf:value "QPFNVTQGKYSTAEECSADSDLNFLIPVAVG" .
<http://rdf.glycoinfo.org/glycan/wurcs/WURCS=2.0/3,4,3/[a2122h-1a_1-5_2*N][a1122h-1a_1-5][a1122h-1a_1-5_6*OP^XOCCN/3O/3=O]/1-2-2-3/a4-b1_b6-c1_c2-d1>
faldo:location <http://natpros.org/peptide#N4loc> .
<http://natpros.org/peptide#N4loc>
faldo:position 4 ;
rdf:type faldo:ExactPosition ;
faldo:reference <http://np.org/seq> .
<http://np.org/seq>
a uniprot:Sequence ;
rdf:value "QPFNVTQGKYSTAEECSADSDLNFLIPVAVG" .
- ガイドラインに従い、identifiers.orgを追加する(http://identifiers.org/ の形式)
<s> rdfs:seeAlso <http://identifiers.org/inchi/InChI=1S/C6H12O6/c7-1-2-3(8)4(9)5(10)6(11)12-2/h2-11H,1H2/t2-,3-,4+,5-,6-/m1/s1> . <s> rdfs:seeAlso <http://identifiers.org/inchikey/WQZGKKKJIJFFOK-VFUOTHLCSA-N> .
SPARQLthon グループ
参考リンク
- これまでの SPARQLthon
参加者
- 片山俊明 (DBCLS)
- 川島秀一 (DBCLS)
- 守屋勇樹 (DBCLS)
- 坊農秀雅 (DBCLS)
- 山本泰智 (DBCLS)
- 河野信 (DBCLS)
- 古崎晃司(大阪大学)
- 山口敦子 (DBCLS)
- 吉沢明康(京都大学)
- 森田 巧(保健同人社)
- 岡別府陽子(MSS)
- 大石直哉(DOGRUN)
- 永野朗夫(PENQE)
- 田中聡 (Trans-IT)
- 大田達郎 (DBCLS)
- 小寺正明(東工大)11日(2日目)のみ参加
- 小澤健太郎 (SGI)
- 上原英也 (SGI)
- 西村悟史 (産総研)
- 信定知江 (NBDC) 10日のみ参加(17時まで)
- 山田一作 (野口研)
- 内藤雄樹 (DBCLS)
- 千葉啓和(基生研)
- 有田正規(遺伝研)
- 仲里猛留 (DBCLS)
- 榊原雄太 (協和発酵キリン) 10日のみ参加
- 小野擁子 (協和発酵キリン) 10日のみ参加
- 櫛田達矢(NBDC)
- 西出浩世(基生研)
- 森宙史(東工大)10日のみ参加
- 藤澤貴智(遺伝研)
- 中村伸朗(JST)