BH13.13/glycodb
提供:TogoWiki
目次 |
糖鎖データベース
糖鎖の連携
メタボロームと糖鎖
メンバー
- 金谷(金谷G) 有田(金谷G) 山田(糖鎖G)櫻井(金谷G)
目的
- これまで糖鎖Gでは、曖昧な構造を含む糖鎖構造を一意の線形文字列で表記できる表記法としてWURCSを開発してきた。WURCSは糖質部分構造のみを文字列化するので文字列一致により同じ糖質が含まれるかを容易に判断することができる。これまでに糖鎖データベース以外に、PDB、ChEBIなどのデータについてWURCSへの変換を行ってきた。今回は、金谷Gの糖質が含まれる可能性のある低分子データ(フラボノイド、KNApSAcK)をMolfileToWURCS(Molfile形式からWURCSへ変換するソフト: java)でWURCSに変換し、どの程度糖質が含まれるか調査・検討する。
フラボノイド データベースデータの中の糖質を見つける
2014/1/27
- 有田さんのフラボノイド(FL)のMolfileをWURCSへ変換(Molfile: 有田→山田)。
- (現時点のWURCS生成プログラムでは、FLの表記法によりMolfileの単糖の種類(立体)を考慮できない問題がある)
- (現時点のWURCS生成プログラムでは、FLの表記法によりMolfileの単糖の種類(立体)を考慮できない問題がある)
2014/1/28 & 30
- フラボノイドIDとWURCSをローカルDBへ登録
- WURCS変換結果:
フラボノイド分子数 5479 WURCSが生成されたフラボノイドID 3246 生成されたWURCS(糖質) 3963 WURCS(糖質)の種類 1435
FL1A1AGM0001 WURCS=1.0/1,0/[1X11Xm|1,5]
FL7AAAGL0052 WURCS=1.0/5,4/[12X2Xh|1,5][1X12Xh|1,5][1X12Xh|1,5][12X2Xh|1,5][1X12Xh|1,5]1+1-2,2+6-1*OCC=^EC(C^ECC^ZCC^ZC$6)/9O/8O*/3=O|2+1-2,5+6-1*OCC=^EC(CC^ECC^ECC^Z$6)/9O*/8O/3=O|3+1-2,4+6-1*OCC=^EC(CC^ECC^ECC^Z$6)/9O/8O*/3=O|4+1,5+2
同一の糖質部分構造を持つフラボノイドデータの例:
ID WURCS
FL5FACGS0045 WURCS=1.0/1,0/[1XX2Xa|1,5]
FL5FFCGS0007 WURCS=1.0/1,0/[1XX2Xa|1,5]
FL3FACGS0068 WURCS=1.0/1,0/[1XX2Xa|1,5|3*OCC/3=O]
↑ 上記単糖に”3*OCC/3=O”が修飾されている構造の例
- フラボノイド分子のMolfileからWURCSが生成されたが、FLのIDをもとにすると糖を含まない分子が475分子含まれていた。
- 例)http://metabolomics.jp/wiki/FL1B19NF0002
- WURCS=1.0/1,0/[UbH|1-2,2:x-1*O(C^ECC^ZCC^ECC^ZCO$7/8$3)/4*|1*O|2:x*OC|3*(CC^ZCCCC$2)]
- 例)http://metabolomics.jp/wiki/FL1B19NF0002
- これらのフラボノイドについて、WURCSにおける対応を今後検討する
- フラボノイド分子のMolfileからWURCSが生成されたが、FLのIDをもとにすると糖を含まない分子が475分子含まれていた。
KAApSAcKデータの中の糖質を見つける
金谷さんのKNApSAcKの5万件(1000分子毎に分割)molデータをMol2WURCSソフトでWURCSへ変換(Molfile: 金谷→山田)
- 結果:
- KS_00Kmol~KS_08Kmol => OK
- KS_09Kmolで以下の分子からWURCS生成に問題がありそう?
- http://kanaya.naist.jp/knapsack_jsp/information.jsp?word=C00009344
- KS_09Kmolは、C00009344以外はOK(~999)
- http://kanaya.naist.jp/knapsack_jsp/information.jsp?word=C00009344
- KS_10Kmol~KS_33Kmol => OK
- KS_34KmolでWURCS生成に時間がかかっているので強制終了。(~52/1000)
- KS_35KmolでWURCS生成に時間がかかっているので強制終了。(~350/1000)
- KS_34KmolでWURCS生成に時間がかかっているので強制終了。(~52/1000)
- KS_36Kmol~KS_50Kmol => OK
- KS_00Kmol~KS_08Kmol => OK
2014/1/29
- KS_34Kmolは、C00034052は2時間30分でWURCS生成できた。
- KS_35Kmolは、C00035350は1時間以内でWURCS生成できた。
- KS_09Kmolの、C00009344は2時間30分でWURCS生成できた。
- KS_34Kmolは、C00034052は2時間30分でWURCS生成できた。
- 結果:KNApSAcKのMolfileでWURCSのルールで糖質とみなす構造をすべてWURCSへ変換することができた。
2014/1/30
- KNApSAcKから生成されたWURCSをローカルDBへ登録(山田)
KNApSAcK分子数 50000? WURCSが生成されたKNApSAcK-ID 17207 生成されたWURCS(糖質) 19623 WURCS(糖質)の種類 11050 分子内にあるWURCSとその個数のパターンの種類(櫻井) 11307
C00040128 WURCS=1.0/9,8/[12211m|1,5][1211h|1,4][adXdXXdm|1*=O|6*C][adXdXXdm|1*=O|6*C][122h|1,4|3*CO|3*O][X212h|1,5][12122h|1,5][12211m|1,5][12112m|1,5]1+1,2+2|2+1,3+5|3+1,4+5|4+1,9+3|5+1,6+3|6+1,8+4|7+1,8+3|8+1,9+2
同一の糖質部分構造を持つKNApSAcKデータの例: KNApSAcK-ID WURCS C00033248 WURCS=1.0/5,4/[1212h|1,5][1211h|1,5][12122h|1,5][12211m|1,5][12112h|1,5]1+1,3+3|2+1,3+2|3+1,5+4|4+1,5+2 C00033249 WURCS=1.0/5,4/[1212h|1,5][1211h|1,5][12122h|1,5][12211m|1,5][12112h|1,5]1+1,3+3|2+1,3+2|3+1,5+4|4+1,5+2 C00041869 WURCS=1.0/5,4/[1212h|1,5][1211h|1,5][12122h|1,5][12211m|1,5][12112h|1,5]1+1,3+3|2+1,3+2|3+1,5+4|4+1,5+2 C00041870 WURCS=1.0/5,4/[1212h|1,5][1211h|1,5][12122h|1,5][12211m|1,5][12112h|1,5]1+1,3+3|2+1,3+2|3+1,5+4|4+1,5+2
2014/1/31
- ChEBIの構造からWURCS生成(山田)
メタボロームデータのアノテーションツールとしての可能性を検討
感染症と糖鎖
メンバー
新町 奥田 山本
目的
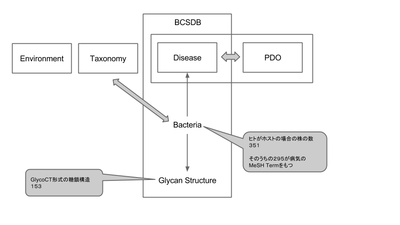
細菌が生産する糖鎖構造の多様性を調べるためのスキームを作成する。
- BCSDB(http://csdb.glycoscience.ru/bacterial/)
- 糖鎖構造(10503 compound)とそのバクテリア菌株(5810 strain)の対応関係
- 菌株名と疾患名との関係
- PDO(http://bioportal.bioontology.org/ontologies/PDO)
- ヒトに感染するバクテリアの感染症のオントロジー
BCSDBとPDOとの連携
- BCSDBの疾患名をPDOのオントロジーにマッピング
2014/1/27、28
- BCSDBから、生物種と糖鎖構造、生物種とその疾患との関係を取得
- BCSDBに登録されている疾患の記述とPDOの記述に対応付けを実施
- 半自動的にプログラムで対応を取った後、手作業ですべてチェック
- BCSDBの疾患名はタイポが多い(20%くらい?)
- 疾患名ではなく、症状も記述されている
2014/1/29、30
- 対応の取れなかった疾患の中からPDOに新規に登録出来るものをチェック(山本)
- 疾患から糖鎖構造を推測するためのSPARQLの開発
- 糖鎖構造153種類(GlycoCT)、バクテリア:295株
- 糖鎖構造が思いの外少ない
2014/1/31
- 各疾患毎の糖鎖構造をSPARQLで取り出すことができた。
- 糖鎖構造のリストから部分構造抽出について検討
病気と生物種(バクテリア)と生物種の持つ糖鎖を取得するクエリ(ホストはヒト):
prefix glyco: <http://purl.jp/bio/12/glyco/glycan/>
prefix dcterms: <http://purl.org/dc/terms/>
prefix bibo: <http://purl.org/ontology/bibo/>
prefix glycodb: <http://purl.jp/bio/12/database/>
prefix uniprot: <http://purl.uniprot.org/core/>
select distinct ?label ?name ?seq ?img
from <http://csdb.glycoscience.ru/integration/bcsdb>
from <http://csdb.glycoscience.ru/integration/image>
from <http://csdb.glycoscience.ru/integration/bcsdb/pdo/mapping>
from <http://purl.bioontology.org/ontology/PDO>
where
{
?o a glyco:source_natural ;
glyco:has_taxon ?tax ;
glyco:hosted_by ?host ;
glyco:has_reference ?ref .
?tax a uniprot:taxon ;
uniprot:scientificName ?name .
?host a glyco:source_natural ;
glyco:has_taxon <http://purl.uniprot.org/taxonomy/9606> ;
glyco:has_disease ?dise .
?dise rdfs:seeAlso ?pdo .
?pdo rdfs:label ?label .
?ref a glyco:referenced_compound ;
glyco:has_glycan ?glycan .
?glycan owl:sameAs ?sameAs .
?sameAs glyco:has_image ?img .
?glycan glyco:has_glycosequence ?gseq .
?gseq glyco:has_sequence ?seq ;
glyco:in_carbohydrate_format glyco:glycoct .
} group by ?label
limit 20
環境微生物と糖鎖
メンバー
新町 奥田
目的
環境中で合成される糖鎖構造の多様性について調べるためのスキーム開発
- 「疾患と糖鎖」で作成したスキームでのPDOの部分をTaxonomyあるいは環境微生物の情報に置き換えて対応
- Taxonomyの階層と糖鎖構造の関係
- 各taxonでの糖鎖構造のプロファイル
- 環境と棲息する微生物種の対応関係(MicrobeDBからなど)
- 環境による糖鎖構造の多様性の違い
- Taxonomyの階層と糖鎖構造の関係
糖鎖構造標準化
脂質
- 脂質の表記方法はある程度決まっているが国際標準化はない(有田さんから)
- 文献に記載されている脂質の構造情報もこれまで蓄積された実験的な事実を考慮して記載されている場合が多いらしい。
糖鎖のRDF化
糖質構造のRDF
メンバー
山田
2014/1/30
- Virtuoso v7をMacにインストール
- Virtuoso へBCSDBのデータを入れた
2014/1/31
- KNApSAcK, Flavonoid Database, ChEBIから生成されたWURCSでトリプル作成
WURCSで用いるURIサンプル http://purl.jp//bio/12/glyco/wurcs/WURCS文字列
- サンプルデータ
sample of triple:
@prefix glyco: <http://purl.jp/bio/12/glyco/glycan/> .
@prefix xsd: <http://www.w3.org/2001/XMLSchema#> .
<http://kanaya.naist.jp/knapsack_jsp/information.jsp?word=C00043421>
a glyco:glycosequence ;
glyco:has_sequence "WURCS%3D1.0%2F1%2C0%2F%5B1111deeA%7C1%2C2%7C4-1%2C8-2%2AOCC%5ESOCC%5ERCNCC%5ERN%2A%2F11C%28C%5EZCC%5EZCC%5EZC%2415%29%2F18OC%2F17Cl%2F10%3DO%2F7C%2F6%3DO%2F4CCC%2F29C%2F3%3DO%7C1%2A%28C%29%7C3%2AC%7C8%2A%3DO%5D”^^xsd:string ;
glyco:in_carbohydrate_format glyco:wurcs .
- Virtuoso へKNApSAcK, Flavonoid Database, ChEBIのデータを入れた
- 全WURCS取得SPARQLクエリ
SELECT ?dburi ?wurcs
WHERE { ?dburi <http://purl.jp/bio/12/glyco/glycan/has_sequence> ?wurcs
FILTER regex(?wurcs, "WURCS") }
- “[12122h|1,5”の部分構造(beta-glucopyranose b-Glcp)を含む糖質取得SPARQLクエリ
SELECT ?dburi ?wurcs
WHERE { ?dburi <http://purl.jp/bio/12/glyco/glycan/has_sequence> ?wurcs
FILTER regex(?wurcs, "%2F%5B12122h%7C1%2C5") }
- beta-glucopyranose (b-Glcp)
を含むDBIDを取得するSPARQLクエリ
http://purl.jp//bio/12/glyco/wurcs/WURCS=1.0/1,0/[12122h|1,5]
SELECT ?dburi
WHERE { ?dburi <http://purl.jp/bio/12/glyco/glycan/has_sequence> <http://purl.jp/bio/12/glyco/wurcs/WURCS%3D1.0%2F1%2C0%2F%5B12122h%7C1%2C5%5D> }
プロテイン名からID
- プロテイン名からUniprotのIDを調べるSPARQL
エンドポイントURL: http://beta.sparql.uniprot.org/
SELECT ?uniprot_id
WHERE
{
?id ?label "ConA" .
?uniprot_id ?p ?id .
?uniprot_id a <http://purl.uniprot.org/core/Protein>
}
/mw/index.php/BH12.12/SPARQLthon/SPARQLendpoints
を参考にする。
back to BH13.13