がんゲノム
提供:TogoWiki
Ryotayamanaka (トーク | 投稿記録) (→Fuseki を使う場合の注意) |
Ryotayamanaka (トーク | 投稿記録) (→Fuseki を使う場合の注意) |
||
| 738行: | 738行: | ||
; COALESCE の変数 | ; COALESCE の変数 | ||
| - | (coalesce(?project_code, "No Data") AS ?project_code) | + | (coalesce(?project_code, "No Data") AS ?project_code) |
のように ?project_code から ?project_code に投影することが Virtuoso では許されるが、Fuseki では以下のエラー | のように ?project_code から ?project_code に投影することが Virtuoso では許されるが、Fuseki では以下のエラー | ||
| 744行: | 744行: | ||
Variable used when already in-scope でエラーになる | Variable used when already in-scope でエラーになる | ||
| - | (coalesce(?project_code, "No Data") AS ?project_code_2) | + | (coalesce(?project_code, "No Data") AS ?project_code_2) |
のように変数名を変更する必要がある | のように変数名を変更する必要がある | ||
2016年2月16日 (火) 08:28時点における最新版
目次 |
がんゲノムとRDFの概要
動機と目的
- がんゲノム・データベースはサンプル(患者さん)の簡易な診断データを含む。
- 将来は医療システム(電子カルテ等)との連携が考えられる。
- がんゲノム・データベースは体細胞変異、遺伝子発現、タンパク発現、Metylation等、多様なOMICSデータを含む。
- ENCODEデータのようなOMICSデータ(BEDファイル・レベルの情報)も今のところRDF化されていない。
- BEDファイル・レベルの情報をRDF化するメリットはあるのか?
- 診断データとOMICSデータをRDF化して統合検索するモデルは作れるか。
- 将来は、電子カルテ(診断)と配列(OMICS)の統合検索が必要になるため、そのひな形になるか。
- これに加えて、公共のRDFデータを加えた検索ができるとさらによい。
- まずは、ICGC Data Portal 同様の検索画面をRDFベースで作れるか。- https://dcc.icgc.org/search
- もしかしたら、Stanza で開発工数を減らせるか。
既存のRDF化プロジェクト
- COSMIC
- D2RQでエンドポイント公開 - リンク
- これは紛らわしい名前だが関係ない - COSMIC SPARQL Endpoint
- TCGA > Linked TCGA
- ICGC > ないようなので作る!
Linked ICGC 概要
サイト
- ICGC --------> https://dcc.icgc.org/
- Linked ICGC --> http://icgc.link/
開発方針
- Linked ICGC データを作って Data Portal の一部を SPARQL ベースで実装
- ICGC Data Portal から Clinical Data と Simple Somatic Mutation のデータをダウンロード
- Donor Data - http://docs.icgc.org/donor-data-file-donor
- Simple Somatic Mutation - http://docs.icgc.org/icgc-simple-somatic-mutation-format
- Mutation の ID 付けなど処理済み - http://docs.icgc.org/methods
- RDFに変換して、Virtuoso にロード
- RDFに変換(詳細は下記)
- Virtuosoにロード、統合するデータもロード、結合用データを生成
- 全てをロード
- SPARQL を発行、D3SPARQLでグラフを描画
- ダウンロードできる表データ
- 表データのRDFへの変換 〜 表の正規化とD2RQ(DirectMapping)
- ダウンロードできるデータは非正規化されている。 例、この3つの表は1つになっている
- そこで、表データのRDF化手順
- 表データをMySQLにロードする。この表は正規化されていない可能性がある。
- 表をMySQL内の他の表にSELECT-INSERTで第3正規形にする。(後に、主キー列が主語で従属列が目的語のトリプルが生成される。)
- D2RQ等でマッピングを生成し、必要に応じてこれを修正する。
データ統合で何ができるか
- 遺伝子を他のデータセットとリンク
- アノテーションの検索、遺伝子変異の多く見られるパスウェイや GO の抽出
- この機能は本家 ICGC データ・ポータルにも実装されている
- WikiPathway など、他のデータセットとも統合が容易といえるか
- アノテーションの検索、遺伝子変異の多く見られるパスウェイや GO の抽出
- 変異を他のデータセットとリンク
- 日本人ゲノム情報との統合 >> 変異から日本人レア・バリアントを除外できる
- ドナーを他のデータセットとリンク
- バイオバンクのドナー情報(+ 標本とサンプルの情報)?
- 電子カルテのドナー情報(+ より詳しい診断情報)?
- 治験のドナー情報?
Linked ICGC 仕組み
サイト
[GitHub] https://github.com/ryotas/linked-icgc
スキーマ
- clinicalデータとsampleデータのオリジナルのスキーマはこれ
- ssm (体細胞変異) データはmutation_idに従属する列と各変異の影響(DNAまたはRNAの置換等)が結合されている
- 正規化された表定義は見当たらないが以下参考
- Primary Analysis - http://docs.icgc.org/simple-somatic-mutations-ssm-primary-analysis-file-p
- Analysis Metadata - http://docs.icgc.org/simple-somatic-mutations-ssm-analysis-metadata-file-m
- 正規化された表定義は見当たらないが以下参考
ICGCデータ
- 二次配布 -- 推奨されている。E9. Data Management Box 8
- "Other portals: The ICGC should encourage the redistribution, integration and visualization of the data by community bioinformatics portals."
- ある変異(例えばMU832963)が同じProjectでのみ高頻度に見つかっている場合がある。これは検出バイアスを示唆するか。このようなケースをクエリできるか。
中継サーバー
クライアントアプリはSPARQLを直接 SPARQL Endpoint(= DB Server)に投げるのではなく、中継サーバーに投げる。
ポータル
ポータルはJavaScriptで書かれたクライアントアプリ
git clone https://github.com/ryotas/linked-icgc-portal cp -r ./linked-icgc-portal/* /var/www/html/
既知の問題
- SSM (Mutation) の検出がされていないサンプルについて、本家ではDonor数に数えられるが、Linkedではクエリ上これが出てこないため(donorとdetectionを結合してしまっている)、そのようなサンプルを含むプロジェクト(BLCA-US, BRCA-UK)ではDonor数が少なく見える
Linked ICGC (データ生成)の手順
21_sed.sh: 空文字をNULLに変換する
ダウンロードされたファイルはタブ区切り形式で、値がない場合は空文字(タブが並んでいる)です。データベースには空文字やゼロではなく、NULLとしてロードしたいため、「\N」で空文字を置換します。
cat clinical.tsv | awk -F"\t" '{for(i=1;i<=NF;i++){if($i==""){$i="\\N"}}OFS="\t";print}' > clinical_sed.tsv
22_mysql.sh: 主キーと外部キー
これらのキーは各 ICGC データを MySQL のデータベースにロードした際に付加しています(22_mysql.sh)。付加することで ICGC からダウンロードしたデータの整合性(IDの一意性、Not NULL、IDの参照)を確認しています。
- project
- PK: project_code
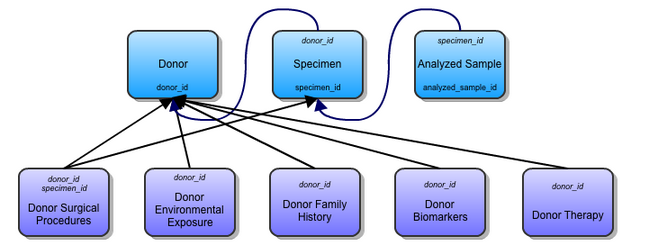
- donor
- PK: icgc_donor_id
- FK: project_code --> project.project_code
- specimen
- PK: icgc_specimen_id
- FK: project_code --> project.project_code
- FK: icgc_donor_id --> donor.icgc_donor_id
- sample
- PK: icgc_sample_id
- FK: project_code --> project.project_code
- FK: icgc_donor_id --> donor.icgc_donor_id
- FK: icgc_specimen_id --> specimen.icgc_specimen_id
- ssm_mutation
- PK: icgc_mutation_id
- ssm_detection
- PK: icgc_donor_id, icgc_specimen_id, icgc_sample_id, icgc_mutation_id
- FK: project_code --> project.project_code
- FK: icgc_donor_id --> donor.icgc_donor_id
- FK: icgc_specimen_id --> specimen.icgc_specimen_id
- FK: icgc_sample_id --> sample.icgc_sample_id
- FK: icgc_mutation_id --> ssm_mutation.icgc_mutation_id
- ssm_effect
- PK: icgc_mutation_id, transcript_affected
- FK: icgc_mutation_id --> ssm_mutation.icgc_mutation_id
ここで主キーの値はリソース URI を作成するために使用することができるため、これに従ってマッピングを定義します。
例:
map:ssm_effect a d2rq:ClassMap; d2rq:class <Effect>; d2rq:dataStorage map:database; d2rq:uriPattern "Effect/@@ssm_effect.icgc_mutation_id@@_@@ssm_effect.transcript_affected@@" . <-- ssm_effect の主キー
23_d2rq.sh: D2RQ でダンプ
- MySQL から D2RQ で RDF データをダンプする
cd d2rq/d2rq-0.8.1 ./dump-rdf -b http://icgc.link/ --verbose mapping-w3c.ttl > $HOME/data/icgc.nt
- この際、性能向上のため、表の JOIN があるとき(= SPO の O が URI であるとき)参照されている列に索引を張っておく
$ vi sql/31_index.sql CREATE INDEX idx_project ON project (project_code); CREATE INDEX idx_donor ON donor (donor_id); CREATE INDEX idx_ssm_mutation ON ssm_mutation (mutation_id);
30_load.sh: Virtuosoへのロード
Virtuosoの設定
- t2.medium(メモリ 4GB)で4GB用の設定にするとデータのロード中になぜか落ちてしまうので2GB用の設定にしておく
- データ作成時には、r3.xlarge(メモリ 30GB)を使い32GB用の設定にしている
./virtuoso.sh edit ;; Uncomment next two lines if there is 2 GB system memory free NumberOfBuffers = 170000 MaxDirtyBuffers = 130000
- ロードするファイルを置くディレクトリを指定する
DirsAllowed = ., /usr/local/share/virtuoso/vad, /home/ec2-user/linked-icgc/rdfize/output
Virtuosoを起動
./virtuoso.sh start
既存のグラフをクリアする
./virtuoso.sh clear http://icgc.link/graph
Linked-ICGCのロード
time ./virtuoso.sh loaddir /home/ec2-user/linked-icgc/rdfize/output *.nt http://icgc.link/graph
Bio2RDFデータのロード
- 引数がよくわからないので loadttl でなく loaddir を使っている
- その場合、quad だがグラフの指定は必要、実際にはこのグラフは作成されず、グラフは「http://bio2rdf.org/hgnc_resource:bio2rdf.dataset.hgnc.R3」となる
time ./virtuoso.sh loaddir /home/ec2-user/data hgnc_complete_set.nq http://hoge/
完了次第、EC2をシャットダウンしたいとき
(time ./virtuoso.sh loaddir /home/ec2-user/data *.nt http://icgc.link/graph > /home/ec2-user/data/icgc.log; sudo shutdown -h now) &
40_construct.sh: 外部データの参照用のトリプルの作成
クエリの実行時間が長いので、タイムアウトしないように設定変更しておく。
$ ~/virtuoso.sh edit ;MaxQueryCostEstimationTime = 400 ; in seconds MaxQueryExecutionTime = 6000 ; in seconds
40_construct.sh を実行してトリプルを作成する。
$ sh 40_construct.sh
40_construct.sh では次のように Construct クエリを実行している。
node ~/sparql-gateway/client.js ./construct/gene.sql ./output/x-gene.nt
生成されたトリプルをロードする。
time ~/virtuoso.sh loaddir ~/linked-icgc/output x-gene.nt http://icgc.link/release_18
50_dump_graph.sh: Virtuosoでグラフをダンプする
dump_one_graph プロシージャを使用してグラフを .ttl ファイルにダンプし、圧縮します。
dump_one_graph プロシージャを作成するため、以下のページのスクリプトを実行します。
次に、50_dump_graph.sh を実行します。
$ sh linked-icgc/50_dump_graph.sh
50_dump_graph.sh の中身は次の通りです。
#/bin/sh!
cd /usr/local/var/lib/virtuoso/db
echo -e "dump_one_graph ('http://icgc.link/release_18','./data_',50000000000);\n" | sh ~/virtuoso.sh isql
データを移動しておきます。
$ mv /usr/local/var/lib/virtuoso/db/data_000001.ttl.gz ~/linked-icgc_20/output/all_projects.ttl.gz
展開して、ロードできることを確認します。(圧縮されたままだとロードできません。)
$ gunzip ~/linked-icgc_20/output/all_projects.ttl.gz $ ~/virtuoso.sh loadttl /home/ec2-user/linked-icgc_20/output/all_projects.ttl http://icgc.link/release_20
上記の処理を通して実行
既存のデータベースを一度削除
~/virtuoso.sh stop ~/virtuoso.sh remove ~/virtuoso.sh start
RDFの作成からロードまで実行
cd ~/linked-icgc sh 10_download_all.sh projects_all.txt sh 20_convert.sh projects_all.txt sh 30_load.sh sh 40_construct.sh sh 50_dump_graph.sh
Linked ICGC (データ生成)の補足
D2RQ Mappingのしくみ
ここでは、D2RQ によって自動的に作成されるマッピングについて見てみます。この自動作成機能は ER モデルをどうやって RDF にマッピングできるかを考える上で興味深いもので、似たものとして W3C 勧告の Direct Mapping があります。今回はこの自動作成を使用して作成したマッピングを手作業で修正しています。将来的には、後述の D2RQ Mapper を使用してマッピングを作成することにしたいと考えています。
D2RQ の自動作成機能は、各テーブルの主キーと外部キーから、リソースとなる列やリソース間の関係性の定義を判別しています。このため、各テーブルが第三正規化されていて、主キーと外部キーた意味的に正しく付与されている必要があります。
主キーを与える
ALTER TABLE ssm_detection ADD PRIMARY KEY (donor_id, mutation_id, specimen_id);
この場合のURIは次のようになる
http://localhost:2020/resource/ssm_detection/donor_id=DO46788;mutation_id=MU102219;specimen_id=SP102622
外部キーを与える
ALTER TABLE ssm_detection ADD FOREIGN KEY (donor_id) REFERENCES donor (donor_id);
そうすることで、表のJOINが可能になる
SELECT DISTINCT * WHERE {
?mutation <http://localhost:2020/resource/ssm_detection#mutation_id> ?mutation_id .
?mutation <http://localhost:2020/resource/ssm_detection#ref-donor_id> ?donor . <-- ref-*** というのができる
?donor <http://localhost:2020/resource/donor#donor_id> ?donor_id .
}
LIMIT 10
- D2RQのDirectMapping作成機能(--w3cオプション)というのは使えていないようだ
- DirectMappingを使用するために他に使えるソフトウェアは?Oracle12cも試したい。
D2RQ Mapperでマッピングを作成する
マッピングファイルの修正
D2RQ Mapper を使ったマッピングを後から手作業で修正します
Class の URI を変更します
- donor/donor_id=XXXXX >> Donor/XXXXX
例:
map:donor a d2rq:ClassMap; d2rq:class <Donor>; d2rq:dataStorage map:database; d2rq:uriPattern "Donor/@@donor.donor_id@@" .
全ての property は vocab/ にします (各表で行名が同じ場合、意味の異なる property が同じ名前になる可能性があることに注意)
- donor#donor_id >> vocab/donor_id
- project#project_code >> vocab/project_code
例:
map:donor_icgc_donor_id a d2rq:PropertyBridge; d2rq:belongsToClassMap map:donor; d2rq:column "donor.icgc_donor_id"; d2rq:property vocab:donor_id .
外部 URI を参照したい場合は URI をベタ書きします
map:ssm_effect_gene_affected a d2rq:PropertyBridge; d2rq:belongsToClassMap map:ssm_effect; d2rq:property vocab:gene_affected; d2rq:uriPattern "http://purl.uniprot.org/ensembl/@@ssm_effect.gene_affected@@" .
本当は以下のように PREFIX を使って書きたかったけれど、
@prefix up-ensembl: <http://purl.uniprot.org/ensembl/> . map:ssm_effect_gene_affected a d2rq:PropertyBridge; d2rq:belongsToClassMap map:ssm_effect; d2rq:property vocab:gene_affected; d2rq:uriPattern "up-ensembl:@@ssm_effect.gene_affected@@" .
これだと以下のように出力されてしまう。残念。
(s) (p) <up-ensembl:ENSG00000068078> .
同じカラムから2つのトリプルは作れない。もしかしたら定義方法があるかもしれないが、この場合は、後からconstructでsameAsのトリプルを作るほうが分かりやすいデータになると考えられる。
つまり、こういう風に書いても、はじめのトリプルしか出力されない。
map:ssm_effect_gene_affected a d2rq:PropertyBridge; d2rq:belongsToClassMap map:ssm_effect; d2rq:property vocab:gene_affected; d2rq:uriPattern "http://purl.uniprot.org/ensembl/@@ssm_effect.gene_affected@@" . map:ssm_effect_gene_affected a d2rq:PropertyBridge; d2rq:belongsToClassMap map:ssm_effect; d2rq:property vocab:gene_affected; d2rq:uriPattern "http://bio2rdf.org/ensembl:@@ssm_effect.gene_affected@@" .
このため、ICGC の名前空間で Gene オブジェクトを作っておいて、
map:ssm_effect_gene_affected a d2rq:PropertyBridge; d2rq:belongsToClassMap map:ssm_effect; d2rq:property vocab:gene_affected; d2rq:uriPattern "Gene/@@ssm_effect.gene_affected@@" .
それと sameAs で UniProt と HGNC にリンクさせる。ついでに、dcterms:identifier も作成しておく。
- construct/gene.sql
PREFIX icgc: <http://icgc.link/vocab/>
PREFIX dcterms: <http://purl.org/dc/terms/>
CONSTRUCT {
?uri_icgc dcterms:identifier ?postfix .
?uri_icgc owl:sameAs ?uri_uniprot .
?uri_icgc owl:sameAs ?uri_bio2rdf .
}
WHERE {
?s icgc:gene_affected ?uri_icgc .
FILTER(!(?postfix=""))
BIND (REPLACE(str(?uri_icgc), "^.*Gene/", "") AS ?postfix)
BIND (IRI(CONCAT("http://purl.uniprot.org/ensembl/",?postfix)) as ?uri_uniprot)
BIND (IRI(CONCAT("http://bio2rdf.org/ensembl:",?postfix)) as ?uri_bio2rdf)
}
【参考】Virtuosoに接続できなくなってしまったとき
このようなエラーメッセージで接続できない
*** Error S2801: [Virtuoso Driver]CL033: Connect failed to 1111 = 1111. at line 0 of Top-Level:
そんなときは、lockファイルを削除して再起動
rm /usr/local/var/lib/virtuoso/db/virtuoso.lck
【参考】SPARQLによるクエリ実行
- 集約関数: COUNT, SUM, MIN, MAX, AVG, GROUP_CONCAT, SAMPLE
- 数値関数: ABS, ROUND, CEIL, FLOOR, RAND
- アンチパターン(Virtuoso)
- これは遅い WHERE { ?s ?p ?o FILTER(STR(?o) = "ENSG00000005187") } -- ?o の索引が使えないからか
- こうした方がいい WHERE { ?s ?p ?o FILTER(?o = "ENSG00000005187" OR ?o = "ENSG00000005187"^^xsd:string) }
【古い情報】データの統合(HGNC)
- 遺伝子のクロスリファレンス / Bio2RDF の HGNC (hgnc_complete_set.nq.gz)
- リテラル(ensemble gene ID)同士を名寄せしなければならないため負荷が大きい。特に型指定が異なることを考慮する必要がある(str()関数を使っている)
- 最も簡潔に統合する方法は次のCONSTRUCTか。ICGCのMutationからBio2RDFのGeneに「ssm_effect:gene_affected_bio2rdf」でつながる。
CONSTRUCT {
?mutation ssm_effect:gene_affected_bio2rdf ?gene_id
} WHERE {
?gene_id bio2rdf_vocabulary:identifier ?id1 .
?mutation ssm_effect:gene_affected ?id2 .
FILTER (STR(?id1) = ?id2)
}
IRI関数を使ってIRIを作ってしまう方法もある。これを使えばon-the-flyでもわりと負荷少なくなるかな?
CONSTRUCT {
?ssm ssm_effect:gene_affected ?iri
} WHERE {
?ssm ssm_effect:gene_affected ?id .
BIND(IRI(CONCAT("http://bio2rdf.org/ensembl:", ?id)) AS ?iri)
}
と思って書いてみたけど、Extimation 402 sec でボツ
SELECT
(?gene_uri AS ?Gene)
(?count AS ?Donors)
WHERE {
{
SELECT
?gene
(COUNT(*) AS ?count)
WHERE {
SELECT DISTINCT
?gene
?donor
WHERE {
?effect ssm_effect:ref-mutation_id ?mutation .
?effect ssm_effect:gene_affected ?gene .
?mutation ssm_mutation:mutation_id ?mutation_id .
?detection ssm_detection:ref-mutation_id ?mutation .
?detection ssm_detection:ref-donor_id ?donor .
}
}
GROUP BY ?gene
}
?gene_uri bio2rdf_vocabulary:identifier ?iri
BIND(IRI(CONCAT("http://bio2rdf.org/ensembl:", ?gene)) AS ?iri)
}
ORDER BY DESC(?Donors)
LIMIT 20
もうINSERTしかない。これだと、Bio2RDFになりensemble gene IDのトリプルは作成されない。結合が目的ならばこれは正しい。
INSERT {
GRAPH <http://icgc.link/graph/insert_01> {
?mutation ssm_effect:gene_affected_bio2rdf ?gene_id
}
} WHERE {
?gene_id bio2rdf_vocabulary:identifier ?id1 .
?mutation ssm_effect:gene_affected ?id2 .
FILTER (STR(?id1) = ?id2)
}
しかし、そういうGeneの分もトリプルを作っておいたほうが結合時にフィルタされる様子がわかりやすそうだ。
INSERT {
GRAPH <http://icgc.link/graph/insert_01> {
?mutation ssm_effect:gene_affected_bio2rdf ?iri
}
} WHERE {
?mutation ssm_effect:gene_affected ?gene .
BIND(IRI(CONCAT("http://bio2rdf.org/ensembl:", ?gene)) AS ?iri)
}
こいつを保存しておいた。
$ ./virtuoso.sh isql SQL> load ./data/insert_01.sql;
【古い情報】データの統合(UniProt)
- UniProt Endpoint: http://beta.sparql.uniprot.org/
- 遺伝子名からタンパク質IDを取ってGOを参照
PREFIX up:<http://purl.uniprot.org/core/>
PREFIX taxon:<http://purl.uniprot.org/taxonomy/>
PREFIX rdfs:<http://www.w3.org/2000/01/rdf-schema#>
PREFIX skos:<http://www.w3.org/2004/02/skos/core#>
SELECT DISTINCT ?protein ?class ?o
WHERE
{
?protein a up:Protein
. ?protein up:organism taxon:9606
. ?protein up:encodedBy ?gene
. ?gene skos:prefLabel "TERT"
. ?protein up:classifiedWith ?class
. ?class rdfs:label ?o
. ?class up:database <http://purl.uniprot.org/database/go>
}
- 現在のHGNCのGeneオブジェクトから参照できる他のID
PREFIX xsd: <http://www.w3.org/2001/XMLSchema#>
PREFIX icgc: <http://icgc.link/vocab/>
PREFIX hgnc_vocabulary: <http://bio2rdf.org/hgnc_vocabulary:>
PREFIX bio2rdf_vocabulary: <http://bio2rdf.org/bio2rdf_vocabulary:>
SELECT DISTINCT
?gene
?donor
?s ?p ?o
WHERE {
?effect icgc:mutation ?mutation .
?effect icgc:gene_affected_bio2rdf ?gene .
?s hgnc_vocabulary:x-ensembl ?gene .
?s ?p ?o .
?mutation icgc:mutation_id ?mutation_id .
?detection icgc:mutation ?mutation .
?detection icgc:donor ?donor .
}
LIMIT 100
- Reactome のProteinオブジェクトからUniProtIDを参照する
Linked ICGC 開発ログ(中継サーバー)
中継サーバーによるキャッシング
- キャッシュサイズの確認
- count: キャッシュの数
- size: 使用サイズ (B), storageSize: 確保されているディスク上のサイズ
- indexSizes.hashQuery_1: インデックスのサイズ(totalIndexSize がメモリに載るようにしたい)
mongo sparql --eval "printjson(db.queryresults.stats())"
- インデックスをメモリにロード
- DB 起動時はデータ/インデックスともにメモリにはキャッシュされていない
mongo sparql
db.runCommand({ touch: 'queryresults', data: false, index: true })
- メモリ使用量
- resident: 物理メモリ使用量 (MB)
- virtual: 仮想メモリ使用量 (MB)
mongo sparql db.serverStatus().mem
- キャッシュのサイズが平均4KB程度 > 10^6 = 1M パターン * 10query * 4KB = 40GB > インデックスは log2(40GB) = 12GB 未満
- パターン数 = プロジェクト 100 * 条件(1-4) 10^4 = 10^6
Linked ICGC 開発ログ(ポータル)
SPARQL Tips
NULLだったら"(No Data)"という文字列を返すようにする
- 例: mutation_cancer_distribution.sql
SELECT DISTINCT
..
(COALESCE(?tumour_subtype, "(No Data)") AS ?Tumour_Subtype)
..
WHERE {
..
OPTIONAL { ?project icgc:tumour_subtype ?tumour_subtype . }
..
NULLの扱いについてはアプリ側でも検討しておくべき。SPARQLの実行結果(例えばJSONで返すとき)にNULLが含まれると、そもそも results の bindings から Tumour_Subtype という要素がなくなる。これを空文字とするような処理が必要か。
{ "head": { "link": [], "vars": ["Project", "Tumour_Type", "Tumour_Subtype", "Donors_Affected"] },
"results": { "distinct": false, "ordered": true, "bindings": [ {
"Project": { "type": "literal", "value": "BOCA-UK" } ,
"Donors_Affected": { "type": "typed-literal", "datatype": "http://www.w3.org/2001/XMLSchema#integer", "value": "7" }
# ここに "Tumour_Subtype" はない
} ] } }
どのエンティティから結合するように書くか
ある条件で数を集計したいエンティティがある場合、そのエンティティから結合するように書くべきだろうか
結合を使い回すために、いつも Donor から結合するようにしている。しかし、クエリの柔軟性は?性能は?
- Donor から結合(facet.sql)
# TRAVERSAL
?donor a <http://icgc.link/Donor> .
OPTIONAL { ?donor icgc:sex ?gender . }
OPTIONAL { ?donor icgc:vital_status ?vital_status . }
#OPTIONAL {
?donor icgc:project ?project .
OPTIONAL { ?project icgc:project_code ?project_code . }
OPTIONAL { ?project icgc:primary_site ?primary_site . }
#}
OPTIONAL {
?donor ^icgc:donor ?detection .
#OPTIONAL {
?detection icgc:mutation ?mutation .
OPTIONAL {
?mutation ^icgc:mutation ?effect .
OPTIONAL { ?effect icgc:consequence_type ?consequence_type . }
OPTIONAL {
?effect icgc:gene_affected ?gene .
}
}
#}
}
- Effect から結合(search_mutations_consequence-type.sql)
# TRAVERSAL
?effect a <http://icgc.link/Effect> .
OPTIONAL { ?effect icgc:consequence_type ?consequence_type . }
OPTIONAL {
?effect icgc:mutation ?mutation .
OPTIONAL {
?mutation ^icgc:mutation ?detection .
OPTIONAL {
?detection icgc:donor ?donor .
OPTIONAL { ?donor icgc:sex ?gender . }
OPTIONAL { ?donor icgc:vital_status ?vital_status . }
OPTIONAL {
?donor icgc:project ?project .
OPTIONAL { ?project icgc:project_code ?project_code . }
OPTIONAL { ?project icgc:primary_site ?primary_site . }
}
}
}
}
d3sparql 変更リクエスト
- 結果セットにNULL値が含まれる場合に空文字を返すようにする
- d3sparql.htmlhash
- before
return {"head": col, "data": data[col].value}
- after
if (data[col] != null) {
return {"head": col, "data": data[col].value}
} else {
return {"head": col, "data": ""}
}
リダイレクト設定
グラフ URI およびリソース URI を解決して「有用な情報」を返せるように、適切な URI にリダイレクトを設定する。
- /etc/httpd/conf/httpd.conf
<Directory "/var/www/html">
AllowOverride All
- .htaccess
RedirectMatch 301 ^/(release.*)$ http://icgc.link/repository/$1/ RedirectMatch 301 Project/(.*)$ http://icgc.link/project.html?project_code=$1 RedirectMatch 301 Donor/(.*)$ http://icgc.link/donor.html?donor_id=$1 RedirectMatch 301 Gene/(.*)$ http://icgc.link/gene.html?gene_id=$1 RedirectMatch 301 Mutation/(.*)$ http://icgc.link/mutation.html?mutation_id=$1
Fuseki を使う場合の注意
Update: 2016-02-16
Oracle 12c を使用する場合はエンドポイントを通常 Fuseki で作成する。
- COALESCE の変数
(coalesce(?project_code, "No Data") AS ?project_code)
のように ?project_code から ?project_code に投影することが Virtuoso では許されるが、Fuseki では以下のエラー
Variable used when already in-scope でエラーになる
(coalesce(?project_code, "No Data") AS ?project_code_2)
のように変数名を変更する必要がある
Linked TCGA
Linked TCGA の概要
- TCGAのデータをRDF化している。RDF化されているのはデータの一部であり、体細胞変異のデータはRDF化されていない。
- RDF化されているデータ: 診断、治療歴、RNA発現、miRNA発現、タンパク発現
- 現在はデータ・タイプで分かれた別々のVirtuosoノードのエンドポイントが癌種10種分のみ公開されている。
- RDFのダンプは癌種は27種分全てが公開されている。http://tcga.deri.ie/dumps/
- と思いきや、これらのダンプはRDF化されたトリプルの一部のようだ(臨床データについてはsampleやpatientもない!)
- 元データとRDFデータの一覧は以下。
- 元のTCGAのデータ一覧 - https://tcga-data.nci.nih.gov/tcga/tcgaDataType.jsp
- Linked TCGAのデータのスキーマ - http://tcga.deri.ie/#schema
- TopFed という Federated query processing をこの Linked TCGA 用に開発している。
- 構成の詳細はここに記載されている。http://www.jbiomedsem.com/imedia/1543416435123551_article.pdf
- 200億トリプルを17(2+6+9)のノードのVirtuoso (オープンソース版)にロードしている。(ページ31)
- ただし、TopFedによる Federation Search のエンドポイントはない。(本当に動くのか?)
- これをセットアップするには多数のVirtuosoノードが必要なためAWSが便利そう、でも検証は見送り。
- LTCGAのチームはPubMedやUniProtを加えた検索の事例を論文にしている。
AWSでロードの性能測定
- Virtuosoへのデータ・ロードには大容量メモリのマシンが望ましいか?
- ダンプは癌種毎で、最大は2959Mトリプル(BRCA)、最小は35Mトリプル(DLBC)
- メモリサイズの異なるEC2メモリ最適化インスタンスでバッファサイズを変更して測定する。
- EC2の場合、大容量メモリでロードして、その後に低料金のインスタンスに変更できる。
- EC2メモリ最適化インスタンス
r3.xlarge (4vCPU, 30.5GiB) $0.420/h r3.2xlarge (8vCPU, 61GiB) $0.840/h r3.4xlarge (16vCPU, 122GiB) $1.680/h r3.8xlarge (32vCPU, 244GiB) $3.360/h
- 2959Mトリプル(BRCA)をロード
- データベース・ファイルの格納にはSSD (300GiB, 最低900-最大3000IOPS) を使用
- 1buffer = 8KB でメモリ使用量を計算(実際には8.5KBくらいらしい)
インスタンス (NumberOfBuffers = メモリ使用量目安) 処理時間 = 処理時間をマイクロ秒から計算 r3.xlarge (2720K = 21.25GB) 45.29h = (159389223+3659429)/1000/60/60 r3.2xlarge (2720K = 21.25GB) 39.18h = (136517653+4547384)/1000/60/60 ※ 性能はバッファサイズに依存していることがわかる。 r3.2xlarge (5450K = 42.50GB) 5.69h = (19529858+946258)/1000/60/60 r3.4xlarge (10900K = 85.00GB) 4.00h = (13322055+1097985)/1000/60/60 r3.8xlarge (21800K = 170.00GB) 3.98h = (12898282+1426560)/1000/60/60 ※ NumberOfBuffers=10900K で充分だったことがわかる。
サンプルSPARQLクエリ
- あるサンプルの情報 - http://digital.path.uab.edu/sparql クエリ結果
PREFIX tcga: <http://tcga.deri.ie/>
PREFIX tcgas: <http://tcga.deri.ie/schema/>
SELECT * WHERE {tcga:TCGA-HT-8010 ?p ?o} LIMIT 100
- 全てのサンプルのID - http://digital.path.uab.edu/sparql
PREFIX tcga: <http://tcga.deri.ie/>
PREFIX tcgas: <http://tcga.deri.ie/schema/>
SELECT * WHERE {
?s a tcgas:patient
. ?s tcgas:tumor_tissue_site ?s
} LIMIT 100
- 全てのサンプルの生存日数(http://digital.path.uab.edu/sparql)
PREFIX tcga: <http://tcga.deri.ie/>
PREFIX tcgas: <http://tcga.deri.ie/schema/>
SELECT * WHERE {
?s ?p tcgas:patient
. ?s days_to_deth
} LIMIT 100
PREFIX tcga: <http://tcga.deri.ie/>
PREFIX tcgas: <http://tcga.deri.ie/schema/>
SELECT * WHERE {tcga:TCGA-HT-8010-g1 ?p ?o} LIMIT 100
- あるサンプルのmiRNA(http://digital.path.uab.edu/sparql)
PREFIX tcga: <http://tcga.deri.ie/>
PREFIX tcgas: <http://tcga.deri.ie/schema/>
SELECT * WHERE {
tcga:TCGA-HT-8010 tcgas:result ?r
. ?r a tcgas:miRNA_result
. ?r togas:reads_per_million_miRNA_mapped ?o
. ?r tcgas:chromosome ?chr
. ?r tcgas:start ?start
. ?r tcgas:stop ?stop
} ORDER BY DESC(?o) LIMIT 100
- あるサンプルの遺伝子発現(http://digital.path.uab.edu/sparql)クエリ結果
PREFIX tcga: <http://tcga.deri.ie/>
PREFIX tcgas: <http://tcga.deri.ie/schema/>
SELECT * WHERE {
tcga:TCGA-HT-8010 tcgas:result ?r
. ?r a tcgas:expression_gene_result
. ?r tcgas:scaled_estimate ?o
. ?r tcgas:chromosome ?chr
. ?r tcgas:start ?start
. ?r tcgas:stop ?stop
} ORDER BY DESC(?o) LIMIT 100