BH16.12/Metabolome
提供:TogoWiki
目次 |
メンバー
- 櫻井、川島(秀一)、川島(武士)、有田
メタボロームチームの目標
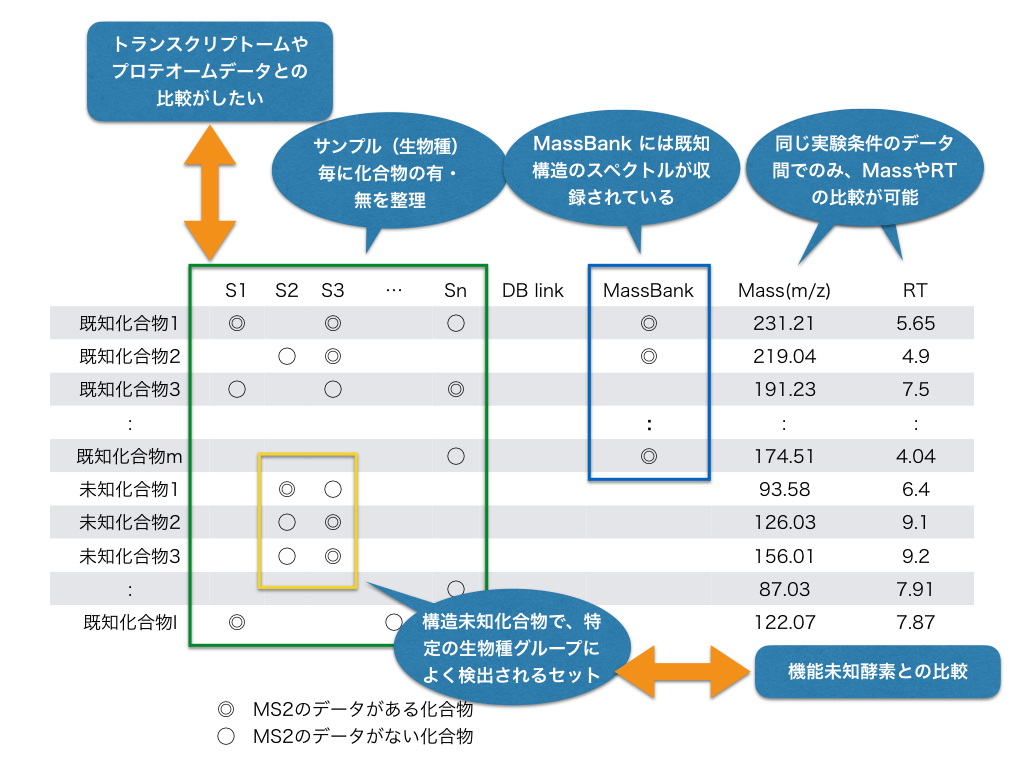
BioMassBank/KomicMarket に登録されているメタボロームデータに関して、生物種別に既知・未知化合物に関しての整理を行う。さらに、整理されたデータをもとにトランスクリプトームやプロテオームのデータと比較解析を行うことができるようなRDF化を行う。
[[1]]
具体的な作業
- Metabolonote に収録されているメタボローム実験のメタデータのRDF化(櫻井)
- KomicMarket, KomicMarket2,
- ビューワーソフトのリリース版準備
- 共通するデータ解析技術で何かコラボ(吉沢さん、高見さん)
- WURCSを付ける検討(糖鎖グループ)
- フラボノイド同定ソフト・化合物名寄せデータベースの原稿修正
- (有田)かずさ野菜メタボロームデータから、共通する代謝物IDの抜き出しと整理
- (有田)TogoMDFormatのJavaパーザー
- MassBank RDF化(川島)
成果
MassBank RDF化
RDF構築
- ドラフトバージョン完成
- 14,240,801トリプル (46,007エントリーから)
- テストエンドポイントにロード
- RDF変換の副産物として、マニュアルに未定義な情報の記載に関して数えあげることができた MassBank未定義タグ
オントロジーマッピング
- SemanticMetabolomics で公開されている、MassBank RDF化のためのオントロジーMBCOについて、今回作成したオントロジーを比較した。
| MassBank ontology | mbco |
|---|---|
| mbo:MassSpectrum | mbco:record |
| mbo:AnalyticalMethod | mbco:mass_spectrometry_assay |
| mbo:MassSpectramMetaData | |
| mbo:SampleChemicalCompound | mbco:chemical_entity |
| mbo:BiologicalSample | NA |
| mbco:mass_spectrum | |
| mbo:has_peak | mbco:has_constituent |
| mbo:mz | mbco:has_intensity |
| mbo:intensity | mbco:has_intensity |
| mbo:relative_intensity | mbco:has_rel_intensity |
SPARQLサンプル
サンプルの生物種別のMassBankエントリーの数
PREFIX mbo: <http://www.massbank.jp/ontology/> SELECT ?name (COUNT(?s) AS ?entry) FROM <http://massbank.jp/> WHERE { ?s mbo:sp_scientific_name ?name } GROUP BY ?name
エントリー毎の実験条件の一覧
PREFIX mbo: <http://www.massbank.jp/ontology/> SELECT ?entry ?column_name ?solvent ?instrument_type ?flow_gradient ?ion_mode FROM <http://massbank.jp/> WHERE { ?entry mbo:analytical_methods_and_conditions ?bn . ?bn mbo:instrument_type ?instrument_type . ?bn mbo:ion_mode ?ion_mode . ?bn mbo:column_name ?column_name . ?bn mbo:solvent ?solvent . ?bn mbo:flow_gradient ?flow_gradient . } LIMIT 100
実験条件の組み合わせでグループ化したMassBankエントリーの数
PREFIX mbo: <http://www.massbank.jp/ontology/> SELECT ?instrument_type ?ion_mode ?column_name ?solvent ?flow_gradient (COUNT(?entry) AS ?massbank) FROM <http://massbank.jp/> WHERE { ?entry mbo:analytical_methods_and_conditions ?bn . ?bn mbo:instrument_type ?instrument_type . ?bn mbo:ion_mode ?ion_mode . ?bn mbo:column_name ?column_name . ?bn mbo:solvent ?solvent . ?bn mbo:flow_gradient ?flow_gradient . } GROUP BY ?instrument_type ?ion_mode ?column_name ?solvent ?flow_gradient
KEGG COMPOUNDと、対応するMassBankエントリー
PREFIX mbo: <http://www.massbank.jp/ontology/> SELECT ?kegg (COUNT(?entry) AS ?massbank) FROM <http://massbank.jp/> WHERE { ?entry mbo:compound ?bn . ?bn a mbo:SampleChemicalCompound . ?bn rdfs:seeAlso ?kegg . FILTER(REGEX(?kegg, "kegg.compound")) } GROUP BY ?kegg
KEGGパスウェイと関連するMassBankエントリーの数
PREFIX rpo: <http://integbio.jp/ontology/> PREFIX rel: <http://integbio.jp/rdf/relation-ontology#> PREFIX mbo: <http://www.massbank.jp/ontology/> PREFIX skos: <http://www.w3.org/2004/02/skos/core#> SELECT ?path ?def (COUNT(?massbank) AS ?massbank) FROM <http://integbio.jp/tmp/> FROM <http://massbank.jp/> WHERE { ?massbank ?p ?sample . ?sample a mbo:SampleChemicalCompound . ?sample rdfs:seeAlso ?cpd . ?path rel:appears ?cpd . ?path skos:definition ?def FILTER(REGEX(?cpd, "kegg.compound")) } GROUP BY ?path ?def
RDF化にあたって今後検討するべきこと
- パーザに一部間違いが見つかったので、FIx する(KEGGへのリンク、PubChemへのリンク)
- 名前空間(オントロジーのURI等)はどうするか
- オントロジーをどのように整備するか
- マニュアルには、Cross-reference to mzOntology: precursor m/z [MS:1000504] のように、mzOntology との対応が意識されている。
- JPOST/HUPO のオントロジーを気にする必要はあるか?
- Mass Spectrometry Ontology http://bioportal.bioontology.org/ontologies/MS もあるがこれはどうなんだろうか?
- マニュアルに記載されていない情報をどう扱うか?
- 記載されていない ION_TYPE
- 記載されていないデータベース
- 記載されていない項目(subtag)
- タイポっぽいのは、修正依頼を出すべき?
- MassBank Wiki に以降する際に解決しそう(有田)
- オントロジーマッピング(SemanticsMetabolomicsと)
- 完了。mbco は、不十分であることが分かった。
- BioMassBank のオントロジーとのマッピング
RDFの利活用
1)興味対象とする生物種で、適切に測定されたメタボロームの測定データを、RDFで検索して集められる。
2)集めた任意の測定データを、アラインメントして、テーブルにする。
3)そのテーブルを軸に、生物種ゲノム vs 代謝物(known/unknown)の解析ができる。食べたものに由来するヒトの代謝物の解析に使える。
1)は、今回のメタデータのRDF化ができたので、可能になる。
2)は、PC上では処理スキームが出来上がっているが、ユーザーが任意に操作するにはまだハードルが高い。KomicMarket2からダウンロードできるTogoMD formatのファイルを使って簡単にアラインメントできるツールを、有田さんが作成中。
3)は、個別研究の中では行えているが、一般ユーザーにはまだハードルが高い。2)を使った簡単な実例を示したい。
メタデータ RDF化
担当:櫻井、川島秀一さん
オントロジー整備(完了)
クラス名はMetabolonoteのxsdをもとに、プロパティ名は、Metabolonoteのプロパティ名をもとにする。
クラス名(大文字始まりのcamel case)、プロパティ名(小文字始まりのcamel caseまたは小文字のsnake case)に合わない部分もあるが、既にMetabolonoteシステムで使われているものがあるので、混乱を避けるため、新しいものは極力作らない。
(例外) D_Recommended_decimal_places_of_m/z ↓ D_Recommended_decimal_places_of_mz ※"/"がQNAMEとして使えなかったため。
RDF整備(完了)
MetabolonoteのAPIをつかってRDFを自動作成するプログラムを作成した。
特定のプロパティについては、よく使われるdctermsやskos、rdf, rdfsのプロパティでの記述を追加した。
(例) SE_Authors -> dcterms:creator *_Comment, *_Comment_of_details -> rdfs:comment *_Descrption -> dcterms:description SE_Reference -> dcterms:references *_Title -> rdfs:label
MSのコメント欄に拡張書式を設定して、カラム、グラジエント、クロマト分離時間をRDFに書き出せるようにした。
[予約語]で始まる行は、行末までを値とする
(今後の拡張)
装置のタイプは、Metabolonoteとその他のシステム(MassBank等)とで意味合いが微妙に異なるので、対応が明確につくものは並記する。 イオン化方法、測定の極性(ポジ・ネガ)は、MassBankの記載方法を並記する。
ツールの公開準備
FlavonoidSearchの投稿完了、N2Dデータベースのウェブサイトの整備完了
糖鎖チームとの連携(計画のみ)
担当:櫻井、山田一作さん
(やりたいこと)
KNApSAcKやKEGGのデータにWURCSで糖鎖情報をつけたものを準備して、化合物アノテーションに付与する。 MS/MSで切り出される可能性がある糖鎖の有無と、切り出されたときのmassを示すことができれば、アノテーションするときにとても便利。
各ピークから、上記情報を用いてGlytoucanへのリンクなどをつけられる。
生物種ごとにWURCSの多様性を見ることができると、遺伝子と統合解析が可能になりそう。
(進捗)
計画のみ完了。
山田さんと、処理スキームについて相談。WURCSを付けるところまでは、既存のjarでできそう。WURCSからmolを起こすこともできそう。C-糖かO-糖かなどの接続様式も判別できそう。
プロテオチームとの連携(相談のみ)
担当:櫻井、吉沢さん、田中さん、河野さん、守屋さん
とりあえずSPLASHを双方でつけてゆくことにする。
UniProt, KEGG pathway等を使って、タンパク-代謝物の情報をつなげることができる。
その他
櫻井作業メモ
2016年12月12日(月)
- これまでの川島さんとの打ち合わせ内容をもとに、以下を実施(櫻井)
- メタデータオントロジーの作成
- Metabolonoteのxsdをもとにowlのオントロジーを整備
- メタデータRDFの修正を開始
- メタデータオントロジーの作成
- フラボノイド同定ソフトの公開準備