BH14.14/compound
提供:TogoWiki
目次 |
化合物関連情報のRDF化
- メンバー: 櫻井、山田、時松、福島、西田、櫛田
- サテライト参加:小寺、木村、三橋、新町、中村
精密質量MSのピークアノテーション(櫻井)
- 精密質量MSのピークアノテーションにおける、イオン化状態(アダクト)の判定アルゴリズムを完成させる
完成させた。
1)指定した上限数の中性の脱離・付加(H2O、アセニト等)、および2)イオン化(プロトン付加、ナトリウム付加等)の組み合わせを解析して、適切なイオンの状態を判別できるようにした。
糖鎖構造のRDF化とSPARQL(山田、新町)
国際糖鎖構造リポジトリGlyTouCanにおいて現在利用している検索ツールは、Glcを検索する場合でもGlcNAcを含んで検索してしまう問題がある(だから今使ってない)。この問題を糖鎖線形表記(WURCS)をRDF化したWURCS-RDFとSPARQLで構造検索を実現したい。 (参考SPARQLthon/glycan/wurcsRDF)
糖鎖構造検索
テスト糖鎖のWURCS ID1 WURCS=2.0/2,3,2/[11122h-1b_1-5][21122h-1a_1-5]/1-2-2/a3-b1_a6-c1 ID2 WURCS=2.0/2,2,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2/a3-b1 ID3 WURCS=2.0/2,2,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2/a6-b1 ID4 WURCS=2.0/2,2,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2/b1-a3|a6 ID5 WURCS=2.0/2,2,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2/b1-a4|a6 ID6 WURCS=2.0/2,2,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2/b1-a3|a4|a6 ID7 WURCS=2.0/2,3,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2-1/a3|a4-b1|c1 ID8 WURCS=2.0/2,3,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2-1/a4|a6-b1|c1 ID9 WURCS=2.0/2,3,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2-1/a3|a6-b1|c1 ID10 WURCS=2.0/2,3,1/[11122h-1b_1-5][21122h-1a_1-5]/1-2-1/b1|c1-a3|a4|a6
構造検索SPARQL作成(山田、新町)
- Exact Substructure match(ESM):グリコシド結合が”曖昧”である場合(wurcs:isFuzzy "true")は”曖昧なグリコシド結合"、”曖昧でない"場合(wurcs:isFuzzy "false")は”曖昧でないグリコシド結合"をそれぞれ検索する方法
- テスト糖鎖10個において、検索できた。
# ID4の糖鎖構造、ESMの条件で検索するSPARQL
PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#>
PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#>
PREFIX owl: <http://www.w3.org/2002/07/owl#>
PREFIX xsd: <http://www.w3.org/2001/XMLSchema#>
PREFIX glycan: <http://purl.jp/bio/12/glyco/glycan#>
PREFIX glytoucan: <http://www.glytoucan.org/glyco/owl/glytoucan#>
PREFIX wurcs: <http://www.glycoinfo.org/glyco/owl/wurcs#>
# SELECT
SELECT DISTINCT ?glycans
str ( ?wurcs ) AS ?WURCS
FROM <http://www.glycoinfo.org/graph/wurcs/0.3.3>
WHERE {
# SEQ
?glycan glycan:has_glycosequence ?gseq
# FILTER
FILTER regex (str(?gseq), "^http://rdf.glycoinfo.org/glycan/") .
# BIND
BIND( iri(replace(str(?glycan), "http://rdf.glycoinfo.org/glycan/", "http://www.glytoucan.org/glyspace/service/glycans/")) as ?glycan2)
BIND( iri(concat(?glycan2, "/image?style=extended&format=png¬ation=cfg"))as ?glycans )
# WURCS
?gseq glycan:has_sequence ?wurcs .
# uniqueRES
?gseq wurcs:has_uniqueRES ?uRES1, ?uRES2.
?uRES1 wurcs:is_monosaccharide <http://rdf.glycoinfo.org/glycan/wurcs/2.0/monosaccharide/11122h-1b_1-5> .
?uRES2 wurcs:is_monosaccharide <http://rdf.glycoinfo.org/glycan/wurcs/2.0/monosaccharide/21122h-1a_1-5> .
# RES
?RESa wurcs:is_uniqueRES ?uRES1 .
?RESb wurcs:is_uniqueRES ?uRES2 .
# LIN
?gseq wurcs:has_LIN ?LINb1a3a6 .
# LIN1
?LINb1a3a6 wurcs:has_GLIPS ?GLIPSb1 , ?GLIPSa3a6 .
# LIN1: GLIPS1
?GLIPSb1 wurcs:has_GLIP ?GLIPb1 .
?GLIPb1 wurcs:has_SC_position 1 .
?GLIPb1 wurcs:has_RES ?RESb .
?GLIPSb1 wurcs:isFuzzy "false"^^xsd:boolean .
# LIN1: GLIPS2
?GLIPSa3a6 wurcs:has_GLIP ?GLIPa3a6 .
?GLIPa3 wurcs:has_SC_position 3 .
?GLIPa3 wurcs:has_RES ?RESa .
?GLIPa6 wurcs:has_SC_position 6 .
?GLIPa6 wurcs:has_RES ?RESa .
?GLIPSa3a6 wurcs:isFuzzy "true"^^xsd:boolean .
}
- Superstructure match(SSM)
- ESM検索のSPARQL文における "?GLIPSb1 wurcs:isFuzzy ..."部分をコメントアウトするだけではダメで、目的の糖鎖構造が取得できなかった。(2/3)
- ESM検索のSPARQL文における "?GLIPSb1 wurcs:isFuzzy ..."と"wurcs:has_GLIP"のFILTERを組み合わせることで、ID2~ID10は目的の検索ができたが、ID1はダメだった。(2/4)
# ESM検索からの変更箇所 # LIN1: GLIPS1 ?GLIPSa3 wurcs:has_GLIP ?GLIPa3 FILTER ( ?GLIPa3 = ?GLIPa3 ) ?GLIPa3 wurcs:has_SC_position 3 . ?GLIPa3 wurcs:has_RES ?RESa . # ?GLIPSa3 wurcs:isFuzzy "fasle"^^xsd:boolean . # LIN1: GLIPS2 ?GLIPSb1 wurcs:has_GLIP ?GLIPb1 FILTER ( ?GLIPb1 = ?GLIPb1 ) ?GLIPb1 wurcs:has_SC_position 1 . ?GLIPb1 wurcs:has_RES ?RESb . # ?GLIPSb1 wurcs:isFuzzy "fasle"^^xsd:boolean .
- ID1をクエリ構造としたSSM検索について、ESM検索のSPARQL文に "MINUS"を書き加えることで、"wurcs:isFuzzy true"を除くことができた。〜ありがとうございました岡別府さん〜(2/4-5)
- GlyTouCanに含まれる36488個の糖鎖(WURCS-RDF)に対してID1の糖鎖構造を検索したところ、約50秒かかった。SAPRQLの改良を検討したい。(山田)(2/5)
- 検索に時間がかかったのは、仮想環境のVirtuosoを利用したからみたい。糖鎖グループのテスト環境で実行したところ、5秒以内には検索できた。(山田)(2/5)
- 今後の方針:入力された糖鎖構造のWUURCS文字列から、構造の特徴(特にwurcs:isFuzzy)を加味したSPARQL文の生成の検討を行う予定。
MINUS {
?gseq wurcs:has_LIN ?LINminus1 .
?LINminus1 wurcs:has_GLIPS ?GLIPSm1 .
?GLIPSm1 wurcs:isFuzzy "true"^^xsd:boolean .
}
- WURCS文字列と検索オプション(検索タイプ、出力情報、LIMITなど)からSPARQLを生成(山田)
- Exact Substructure match(ESM)を実行するSPARQLはできた。
- 他については、SPARQLの書き方が決まったらやる予定。
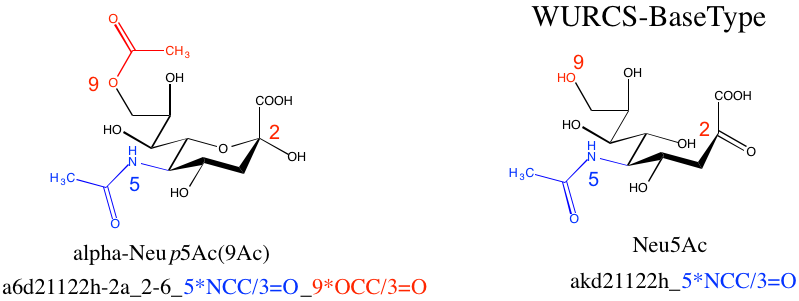
糖鎖構造検索のための基本単糖(WURCS-Basetype)の検討(山田)(2/5~6)
- 目的:糖鎖の領域では単糖が何なのか明確でないところがある。基本的にはポリヒドロキシアルデヒドやポリヒドロキシケトンおよびその誘導体として理解されているが、GlcNAcはGlcの誘導体と考えるよりは、別の単糖であるとすることが一般的なようである。しかし、硫酸化単糖の硫酸化部分は単糖の修飾と考えられることがある。このような曖昧な部分を基本単糖(WURCS-Basetype)を定義して明確としたい。
- WURCS-Basetype:WURCSで定義する基本単糖のこと(まだ脳内レベル)。
- alpha-Neu5pAc(9Ac) をWURCSの構成要素であるuniqueRESで表すと”a6d21122h-2a_2-6_5*NCC/3=O_9*OCC/3=O” となる。これは、アノマーや9位の修飾情報などを含んでいる。しかし、単糖の種類はN-アセチルノイラミン酸である。
- N-アセチルノイラミン酸のWURCS-Basetypeは”akd21122h_5*NCC/3=O”となる。これは以下の処理によりuniqueRES”a6d21122h-2a_2-6_5*NCC/3=O_9*OCC/3=O” から生成できる。
- 削除:アノマー(2a)
- 削除:環(2-6)
- 削除:9位の修飾(9*OCC/3=O)
- 置換:SkeletonCode内のアノマー位の立体情報(6)をケトン”k”へ(アルドースの場合は”o”)
- 置換:修飾によりSkeletonCodeの文字が標準から異なっている場合など(例えば,”A”->”a”)
- しかし、5位のNAcは、ノイラミン酸の炭素骨格に結合している酸素原子がNAcに置換したものであるため残す。
- このような処理をGlyTouCanに含まれる36488個の糖鎖に対して適用したところ、まだ問題点はあるが 652個のWURCS-Basetypeがえられた。
- WURCS-Basetypeでは、基本となるポリヒドロキシアルデヒドやポリヒドロキシケトンの炭素骨格に結合している酸素、水素が置換されている場合は、該当する修飾は残すことにしている。この処理で、GlcNAcがGlcとなってしまうことが防ぐことができる。また、デオキシ糖(Fucなど)も同様にWURCS-Basetypeとしてカウントされる。
WURCS-baseTypeの一例 a1122h a1122h_2*N a2112h_4*NCC/3=O_5*NCC/3=O h1112h h1122h_2*N h1122h_2*NCC/3=O o1111h o1121a_2*NCC/3=O_3*NCC/3=O o1122h_2*B o1122h_2*NCC/3=O o2122h_2*NCC/3=O_4*F o2122h_2*NCC/3=O_4*NCC/3=O o2122h_2*NCC/3=O_6*Cl o2122h_2*NSO/3=O/3=O
糖鎖構造のRDF化(山田)
- WURCS to WURCS-RDF修正: 糖鎖構造の線形文字列表記(WURCS)を、WURCS-RDF形式へ変換するツールの改良
- GlyTouCanに含まれる36488個の糖鎖をWURCS-Basetypeのトリプルを加えてWURCS-RDFを作成。トリプル数:6098496(山田)(2/6)
<http://rdf.glycoinfo.org/glycan/GxxxxxMS/wurcs> wurcs:has_basetype <http://rdf.glycoinfo.org/glycan/wurcs/2.0/basetype/o1221m> ; wurcs:has_basetype <http://rdf.glycoinfo.org/glycan/wurcs/2.0/basetype/o2112h> ; wurcs:has_basetype <http://rdf.glycoinfo.org/glycan/wurcs/2.0/basetype/akd21122h_5*NCC/3=O> ;
天然物の化学情報等のRDF化(時松、山田、櫻井、小寺、西田)
既存vocabulary
天然物(アルカロイド)骨格分類データベース
- 天然物骨格DBに骨格構造をMolfile(V2000)形式で保存、このDBのIDを仮IDとして利用する。(2/3)
- chemdoodleを利用した骨格の化学構造表示に一部変更~結合表示等の問題解決のため~(2/4)
- 今後、化学構造(糖鎖構造)をSVGで表示するStanza:JavaScriptを作りたい。(山田)
- D3.jsを利用する予定 参考サイト Graceful Tree Conjecture, OrgoShmorgo
- 今後、化学構造(糖鎖構造)をSVGで表示するStanza:JavaScriptを作りたい。(山田)
天然物オントロジー
- 骨格の階層分類の方針:天然物(アルカロイド)の生合成をデータ化しながら、骨格を抽出して階層について検討する。
- ChEBIよりもDNP(Dictionary of Natural Products)に似た構造になると考えている。
- 参考
- SPARQLThon/天然物
- SPARQLThon20化合物の中には複数のコンポーネントから生合成されているものがある。糖脂質(糖鎖+脂質)、アルカロイド+テルペノイド、といった化合物を RDF でどう記述するか(記述するのか)を検討
天然物生合成RDF
- 骨格の生合成データベースの構築
- まだたたき台の議論中で方向性が見えてきたところ
- 下記4点のことを今後やる方向で
- 二次代謝産物骨格の収集とデータベース化
- 収集した代謝産物骨格の階層分類
- 代謝産物骨格の生合成データベース化
- アトムトレース
- PIERO reaction ontology 水面下でアップデート中(小寺)
- PIEROのttlのbulkload方法
- 現在、化合物変換を表す語彙(dehydrogenation, hydro-addition, etc)と実際の酵素反応との対応関係を整理しています(小寺)
- 化合物を構成する化学構造については、KCF-S を用いた自動アノテーションを検討しています(小寺)
- (mass profile databaseなどに含まれるpathwayとの関連付けが望まれる)metaboliteでKEGGのIDと1対1の対応が付けることができないcompoundは多く存在する。自動アノテーションが実現されればこの問題の解決の第一歩となる。1対1の対応でなくてよいので「pathwayの大体どのあたりか」でよいので自動で対応付けを行ってくれる機能が求められている (expertでない者はそのような対応付けを行うことができない)。
- なるほど、化合物中の部分構造を自動生成して、それに対応するパスウェイをリストアップするようなことが出来れば良いということでしょうか?KEGG pathwayとの対応関係でしたら、できるかも知れません。試してみます(小寺)
- 自動生成とは独立に生合成知識ベースでの骨格のデータベース化と階層分類(Ontology?)は必要と思いますがどうでしょう(時松:作業中)
- はい、必要だと思います。知識ベースのほうは、ぼくはしばらく手を出せないと思いますので、よろしくお願いします。将来的には、それとKCF-Sとの対応関係も整理できると良いですね。(小寺)
- はいほぼ仰る通りです。KEGG pathwayとの対応関係が知れると非常に助かります。KNApSAcK compound および KEGG DRUGとの関係もわかるようであれば研究の幅が確実に広がると思います。(最悪の場合)名前しか情報として持たないようなmetaboliteのlistからでもpathwayをlistupしてくれるような機能があると助かる、と考えています。 (西田)
- PubChem REST で”aspirin”を調べてみる。これで、aspirinのInChIや他のDBのID、シノニムが取得できるので色々使えるのでは。しかし、取得できたIDがどのDBのIDかの情報はないので注意。また、NBDC版日化辞では日本語でも検索できるようになる予定です。(山田)(2/5)
- 情報ありがとうございます。確かにこのPubChem RESTは押さえておくべきですね (西田)
- 化合物構造を枝刈りし母核と枝の部分構造群(アトムトレース情報有)をつくって、クラスタリングなどを行い使っていく化合物の母集団について検討しては如何でしょう?(山田)
- 母核を共有する化合物グループを定義していただければ、共通するKCF-S部分構造との対応を調べてみることは可能と思います(小寺)
- KCF-Sの最新情報をタブ区切りファイルにまとめました(小寺)
- kcfs2count_v0.3.txt.zip KCF-S のIDとその意味
- kcfs2map_v0.3.txt.zip KCF-S とKEGG pathway mapとの関係
- kgcpd_kcfsd_v0.3.txt.zip KCF-S と KEGG compound との関係
- kgdrg_kcfsd_v0.3.txt.zip KCF-S と KEGG drug との関係
- knapsack_kcfsd_v0.3.txt.zip KCF-S と KNApSAcK との関係
- 参考
アトムトレース:今後の検討項目
- 化合物アラインメントによる自動計算結果を蓄積することはできないでしょうかね?(小寺)
- 蓄積方法を考えたいですね。原子レベルに加えて、生合成のビルディングブロックのトレースを蓄積、表示できる方法がほしいなと(時松)
- 原子レベルで化学構造とトレース情報をRDFで蓄積するのはどうですか?そのために、SIO,ChemInfなどを調査する予定。KCF-Sの拡張は可能?(山田)(2/4-5)
- SIO,CHEMINFともに、分子の元素記号、電荷、結合を記述するのはなさそうだ。(山田)(2/5)
- KEGG RPAIR には、手作業でチェックした atom-to-atom mapping の情報はあります。RDFでどう表現するかという問題はありますが、いちおうデータソースとして使えると思います。原子レベルではなく部分構造レベルにしようと思ったら、どうすればいいか現在よく分かりませんけどね。(小寺)
- RPAIR は当然念頭にあるのですが、KEGGにない代謝産物骨格をかなり追加する必要があるので、それらについては E-zymeなどで atom-to-atom mapping を取る必要があります。今のところ、作成した代謝産物骨格については手作業で生合成ユニットの由来情報をメモしています(atom-to-atomまではしていません)。(時松)
- なるほど、そうですよね。では、(1)反応が既知であるか否かに関わらず、関係のある化合物ペアを集めて、(2)SIMCOMP等でatom-to-atom mappingを計算して、手作業で直したものを集めるという流れでやることになるんでしょうね。手作業で直そうと思ったら SIMCOMPよりは E-zymeのオプションでやるほうがやりやすいかも知れませんが。ひとまず(1)だけでも大仕事になりそうですね。(小寺)
- 方向性はそんな感じかと思います。今のところ、まだ整理の段階で手作業で構造式描画ソフトなどで頭を整理している段階です。上記の作業をするためのワークフローについては、今後SPARQLthonで詰めていきたいと思います。(時松)
- 上にリストアップしたタブ区切りファイルは、KCF-Sと原子との対応関係を表現したものではありません。しかし、原子との対応関係を含めたデータもあるにはあります。RDFでどう表現するかという問題は解決されていません。(小寺)
- トレース情報を化学構造に重ねて表示するなどの機能を持ったStanzaを作りたい。(山田)(2/4-5)
- どんなイメージでしょうか?たとえば、入力化合物を指定して、5ステップ後の化合物(最終生成物)を指定すれば、最終生成物中にある、入力化合物由来の原子が赤く表示されるという感じでしょうか?(小寺)
- (最終)生成物中にある、由来化合物の原子を由来毎に色分けして化学構造を表示させるような機能があるStanzaを作りたいと思っています。どのように色分けするかとかまでは、まだ考えていません。(山田)(2/6)
- どんなイメージでしょうか?たとえば、入力化合物を指定して、5ステップ後の化合物(最終生成物)を指定すれば、最終生成物中にある、入力化合物由来の原子が赤く表示されるという感じでしょうか?(小寺)
KEGG compound と KEGG drugの同一entryが知れるようにしてほしい(Remark fieldがSame as…となっているもの) (2/3)
- LinkDBには、KEGG内部のIDリンク情報がない。しかし、KEGG compound と KEGG drugは日化辞IDとつながっている。以下のRDFを取得すればできそう。
- from "Compound" to "NIKKAJI" in format "turtle" -> download
- from "Drug" to "NIKKAJI" in format "turtle" -> download
- NBDC版日化辞RDF(下記参照)が公開されれば、可能になる予定。
ChEBI ontologyとcompoundのlinkができるような何か(2/3)
- LinkDBで同様に
- from "Compound" to "ChEBI" in format "turtle" -> download
化学構造式エディタ〜化学構造式のデータを作成したい時の参考〜
KEGG PATHWAYを補完したpathway上でのmetabolome profileの可視化(西田、福島、時松)
(動機、目的) Plant metabolome profile databaseの全て情報を pathway 上で可視化したい
- KEGGのcompound IDとの1対1の対応付けでは同定したmetaboliteの半数ほどしか関連付けができない
- KEGG以外のpathway databaseを参照しても、二次代謝産物の多くはそれらと関連が付かない。
- KNApSAcK は「化合物」databaseとしては上記pathway databaseで関連が付かない情報の多くが参照できるが「omics情報を可視化するためのpathway network」情報を持たない
- しかしながら関連付けができないmetaboliteも「pathwayの大体どのあたりにassignすればよいか」がわからないわけではない
- 「expertが目で確認すれば」できないわけではない
- これを自動で行うsystemの確立がしたい
- RDF活用方法も調査、検討 (前節「天然物の化学情報等のRDF化」の実用例)
既存のreferenceとなるpathway databaseの調査
- KEGG
- plant secondary metabolism, flavonoid系pathway(2つ)でもpathwayに対応付けられていないflavonoidが多数
- AraCyc
- KEGGにassignできないcompoundはほぼAraCycにもassignできない
- open linked dataを提供していない。pathway情報を記録したbiopaxがRDFとしては入手可能だが、登録制でIRI(URI)はそのbiopax内でしか使えない。
- AraCycはsuperpathwayの集合で俯瞰しやすいpathwayを持たない http://pmn.plantcyc.org/PLANT/NEW-IMAGE?type=PATHWAY&object=PWY-5320
- plantReactome
- http://plantreactome.oicr.on.ca/entitylevelview/PathwayBrowser.html flavonoid pathwayを例に取るとpathway入口の数反応程度しかcoverしていない
(現状行えることとして) マニュアルキュレーションでmetaboliteをmap, assign, layoutしたpathwayのWikipathwaysでの公開
- BH14.14中でのpathway RDFの利用は難しい
- 何故Wikipathwaysか
- 試験的ではあるがWikipathwaysで公開したpathwayのRDF dumpが行われている (http://www.wikipathways.org/index.php/Help:WikiPathways_RDF)
- prefixには [identifiers.org/cas, pubmed, biopax, identifiers.org/ncbigene], wikipathwaysのxml(GPML)のvisualization用vocabularyが用いられている。
- Userが自由に改変可能なpathway RDF
- 試験的ではあるがWikipathwaysで公開したpathwayのRDF dumpが行われている (http://www.wikipathways.org/index.php/Help:WikiPathways_RDF)
- AtMetExpressにある化合物ID対応のマニュアルキュレーション(時松)
- AtMetExpress 136 MassPeak のデータの KEGG COMPOUND ID/KNApSAcK データのマニュアルキュレーション
- 手作業とPubChem RESTのSynonym利用と二方法を組み合わせて作業
- 136ピークのうち、102ピークについてKEGG COMPOUND ID 付与可能(うち81ピークは単一化合物、重複除き62化合物)
- KNApSAcK 構造式修正 1件 Feedback、LinkDB リンク追加 2件 FeedBack
- フラボノイドに複数の修飾官能基がある化合物など複雑な二次代謝産物については、現状データベース側のSynonym表記の問題でMetMaskでIDを拾いきれてない状況がありそう。
- AtMetExpressで複数の化合物がアノテーションされていて、現状マッピングすべき代謝産物が決定できないピークについては保留して次のステップへ
- AtMetExpress 136 MassPeak のデータの KEGG COMPOUND ID/KNApSAcK データのマニュアルキュレーション
- AtMetExpress(62代謝産物)、KNApSAcK Arabidopsis 代謝産物(626代謝産物)の代謝産物のKEGG PATHWAYでの分布の調査
- KNApSAcK 626代謝産物のうち、LinkDBでKEGG COMPOUND ID がついたものが 446 代謝産物、PubChem REST の Synonym検索で 39 代謝産物を追加、計 485 代謝産物がKEGG ID 付与可能。
- 上記で見たAtMetのデータは理研測定データのSubsetなので、測定可能化合物データについて検討中。会期内には終了しないので継続作業として持ち越し。
PathVisio(Wikipathways読み書きsoftwarel), Wikipathways RDF
- Wikipathways 専用clientとしてPathVisioがある http://www.pathvisio.org/
- Wikipathways の vocabulary (http://vocabularies.wikipathways.org/ )はwpとgpmlに大別されている。gpmlにlayoutなど見栄えに関する情報がある。
NBDC版日化辞RDFデータの作成と提供準備(木村,中村,三橋@JST(東京)、櫛田)
NBDC版日化辞RDFデータの作成(J-Global版RDFデータの修正、追加)
最新のNBDC版日化辞RDFデータ例(抜粋)
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
@prefix rdfs: <http://www.w3.org/2000/01/rdf-schema#> .
@prefix owl: <http://www.w3.org/2002/07/owl#> .
@prefix foaf: <http://xmlns.com/foaf/0.1/> .
@prefix dc: <http://purl.org/dc/elements/1.1/> .
@prefix dct: <http://purl.org/dc/terms/> .
@prefix schema: <http://schema.org/> .
@prefix dbpo: <http://dbpedia.org/ontology/> .
@prefix vcard: <http://www.w3.org/2006/vcard/ns#> .
@prefix bibo: <http://purl.org/ontology/bibo/> .
@prefix prism: <http://prismstandard.org/namespaces/basic/2.0/> .
@prefix tl: <http://purl.org/NET/c4dm/timeline.owl#> .
@prefix jst: <http://vocab.jst.go.jp/terms/sti#> .
@prefix jstd: <http://vocab.jst.go.jp/terms/vlist#> .
@prefix jstpat: <http://vocab.jst.go.jp/terms/patent#> .
@prefix ndl: <http://ndl.go.jp/dcndl/terms/> .
<http://stirdf.jst.go.jp/id/200907000000088846>
rdf:type jst:Chemical ;
jst:nikkaji-ID-registry "02722998K" ;
rdfs:label "4‐(トリイソプロピルシリルオキシ)フラン‐2‐カルボアルデヒド"@ja, "4-(Triisopropylsilyloxy)furan-2-carbaldehyde"@en ;
foaf:depiction "http://api.jglobal.jst.go.jp/chemical/images/272/J2.722.998K.gif" ;
jst:nikkaji-number "J2.722.998K" ;
dc:identifier "J2.722.998K" ;
owl:sameAs <http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K> ;
jst:chem-formula "C<sub>14</sub>H<sub>24</sub>O<sub>3</sub>Si", "C14-H24-O3-SI" ;
jst:molar-weight-min "268429" ;
jst:molar-weight-max "-1000".
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K>
jst:InChI "InChI=1S/C14H24O3Si/c1-10(2)18(11(3)4,12(5)6)17-14-7-13(8-15)16-9-14/h7-12H,1-6H3" ;
jst:InChIKey "JIUJGYWGJHLBIJ-UHFFFAOYSA-N" ;
<http://semanticscience.org/resource/CHEMINF_000200> <http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi>,
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi_key>.
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi>
<http://semanticscience.org/resource/SIO_000300> "InChI=1S/C14H24O3Si/c1-10(2)18(11(3)4,12(5)6)17-14-7-13(8-15)16-9-14/h7-12H,1-6H3";
rdf:type <http://semanticscience.org/resource/CHEMINF_000113>.
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi>
<http://semanticscience.org/resource/SIO_000300> "JIUJGYWGJHLBIJ-UHFFFAOYSA-N";
rdf:type <http://semanticscience.org/resource/CHEMINF_000059>.
<http://semanticscience.org/resource/CHEMINF_000200>
rdfs:label "has attribute".
<http://semanticscience.org/resource/SIO_000300>
rdfs:label "has value".
<http://semanticscience.org/resource/CHEMINF_000113>
rdfs:label "InChI descriptor".
<http://semanticscience.org/resource/CHEMINF_000113>
rdfs:label "InChIKey".
修正・追加の内容(2/2-5)
- 1.Molfile(構造画像)データのpredicate修正 (foaf:image → foaf:depiction)
- 2.dc:identifier "ID番号" の追加 (dc:identifier "J2.722.998K") ←RDFガイドラインに対応
- 3.個々の化合物に rdf:type jst:Chemical がついていることを確認 ←RDFガイドラインに対応
- 4.SIO, CHEMINFを使ったInChI, InChIKey情報のRDFデータ作成
J-Global版日化辞RDFデータのInChI,InChIKey情報の記述例
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K>
jst:InChI "InChI=1S/C14H24O3Si/c1-10(2)18(11(3)4,12(5)6)17-14-7-13(8-15)16-9-14/h7-12H,1-6H3" ;
jst:InChIKey "JIUJGYWGJHLBIJ-UHFFFAOYSA-N" .
↓↓↓
NBDC版日化辞RDFデータのInChI,InChIKey情報の記述例(SIO, CHEMINFを使用)
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K>
jst:InChI "InChI=1S/C14H24O3Si/c1-10(2)18(11(3)4,12(5)6)17-14-7-13(8-15)16-9-14/h7-12H,1-6H3" ;
jst:InChIKey "JIUJGYWGJHLBIJ-UHFFFAOYSA-N" ;
<http://semanticscience.org/resource/CHEMINF_000200> <http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi>,
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi_key>.
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi>
<http://semanticscience.org/resource/SIO_000300> "InChI=1S/C14H24O3Si/c1-10(2)18(11(3)4,12(5)6)17-14-7-13(8-15)16-9-14/h7-12H,1-6H3";
rdf:type <http://semanticscience.org/resource/CHEMINF_000113>.
<http://stirdf.jst.go.jp/cde/nikkaji/J2.722.998K_standard_inchi_key>
<http://semanticscience.org/resource/SIO_000300> "JIUJGYWGJHLBIJ-UHFFFAOYSA-N";
rdf:type <http://semanticscience.org/resource/CHEMINF_000059>.
<http://semanticscience.org/resource/CHEMINF_000200>
rdfs:label "has attribute".
<http://semanticscience.org/resource/SIO_000300>
rdfs:label "has value".
<http://semanticscience.org/resource/CHEMINF_000113>
rdfs:label "InChI descriptor".
<http://semanticscience.org/resource/CHEMINF_000059>
rdfs:label "InChIKey".
- 5.smiles情報の追加
- OpenBabel2.3.1を使ってSDファイルからcanonical Smilesを生成(下記を参照したBH12.12/UT Prot)。
コマンド
obabel *.sd -ocan
入力ファイルの例
03000001C
JEMaster102D
JICST Standard molfile 1.0
36 38 0 0 1 0 0 0 0 0 1 V2000
6.4489 0.3672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
5.4641 0.1936 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 c 1 0 0
7.4337 0.1936 0.0000 S 0 0 0 0 0 6 0 0 0 0 0 0 c 0 0 0
6.2753 1.3520 0.0000 S 0 0 0 0 0 6 0 0 0 0 0 0 c 0 0 0
6.6226 -0.6176 0.0000 F 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
5.4641 -0.8064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
4.5980 0.6936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
5.5718 0.8041 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
8.4185 0.0199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
7.6074 1.1783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
7.2601 -0.7912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
6.1016 2.3368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
5.2905 1.1783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
7.1413 1.8520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
4.5980 -1.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
6.3301 -1.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
3.7321 0.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
4.5980 1.6936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
8.7605 -0.9198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
9.0612 0.7860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
6.8677 2.9796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
5.1619 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
4.5980 -2.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
6.3301 -2.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
2.8660 0.6936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
3.7321 -0.8064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
9.7454 -1.0934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
10.0460 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
6.6940 3.9644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
4.9883 3.6637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
5.4641 -2.8064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
2.0000 0.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
10.3881 -0.3274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
5.7543 4.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 r 0 0 0
5.4641 -3.8064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
4.5980 -4.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 c 0 0 0
1 3 1 0 0 0 0
1 4 1 0 0 0 0
1 5 1 0 0 0 0
2 1 1 6 0 0 0
2 6 1 0 0 0 0
2 7 1 6 0 0 0
2 8 1 0 0 0 0
3 9 1 0 0 0 0
3 10 2 0 0 0 0
3 11 2 0 0 0 0
4 12 1 0 0 0 0
4 13 2 0 0 0 0
4 14 2 0 0 0 0
6 15 2 0 0 0 0
6 16 1 0 0 0 0
7 17 1 0 0 0 0
7 18 2 0 0 0 0
9 19 2 0 0 0 0
9 20 1 0 0 0 0
12 21 2 0 0 0 0
12 22 1 0 0 0 0
15 23 1 0 0 0 0
16 24 2 0 0 0 0
17 25 1 0 0 0 0
17 26 2 0 0 0 0
19 27 1 0 0 0 0
20 28 2 0 0 0 0
21 29 1 0 0 0 0
22 30 2 0 0 0 0
23 31 2 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
27 33 2 0 0 0 0
28 33 1 0 0 0 0
29 34 2 0 0 0 0
30 34 1 0 0 0 0
31 35 1 0 0 0 0
35 36 1 0 0 0 0
M END
><JICST_ID>
03000001C
$$$$
03000002A
JEMaster102D
JICST Standard molfile 1.0
...
出力ファイルの例
COC(=O)C(=C)[C@H](C(S(=O)(=O)c1ccccc1)(S(=O)(=O)c1ccccc1)F)c1ccc(cc1)OC 03000001C
結果 → 日化辞の2,514,562化合物のSmilesを生成
今後の追加予定
- APIの実装(PUG RESTを参考)
- smiles, CAS登録番号情報のRDF化(SIO, CHEMINFに準拠、CAS登録番号は内部のみで使用)
- ChemSpider, ChemIDplus等のリンク情報の追加(rdfs:seeAlso)
- ガイドラインへの対応(rdf:type, rdfs:label, rdfs:range, rdf:domainなど)
サンプルRDF
<http://stirdf.jst.go.jp/cde/nikkaji/J367.842C>
<http://semanticscience.org/resource/CHEMINF_000200> <http://stirdf.jst.go.jp/cde/nikkaji/J367.842C_cas_registry_number>.
<http://stirdf.jst.go.jp/cde/nikkaji/J367.842C_canonical_smiles>.
<http://stirdf.jst.go.jp/cde/nikkaji/J367.842C_cas_registry_number>
<http://semanticscience.org/resource/SIO_000300> "93966-58-6";
rdf:type <http://semanticscience.org/resource/CHEMINF_000446>.
<http://stirdf.jst.go.jp/cde/nikkaji/J367.842C_canonical_smiles>
<http://semanticscience.org/resource/SIO_000300> "CCCCOCC(CNc1ccc(cc1)Cc1ccc(cc1)N)O";
rdf:type <http://semanticscience.org/resource/CHEMINF_000007>.
<http://semanticscience.org/resource/CHEMINF_000200>
rdfs:label "has attribute".
<http://semanticscience.org/resource/SIO_000300>
rdfs:label "has value".
<http://semanticscience.org/resource/CHEMINF_000446>
rdfs:label "CAS registry number".
<http://semanticscience.org/resource/CHEMINF_000007>
rdfs:label "canonical SMILES descriptor".
<http://stirdf.jst.go.jp/cde/nikkaji/J367.842C>
rdfs:seeAlso <http://rdf.chemspider.com/search/HBHGSQLFKAKHML-UHFFFAOYSA-N>,
<http://chem.sis.nlm.nih.gov/chemidplus/rn/93966-58-6>.
トリプルストアの準備(三橋@東京)
- virtuosoのインストール完了、起動確認。
- データのアップロード