BH14.14/Visualization
提供:TogoWiki
(版間での差分)
(→Visualize) |
(→Visualize) |
||
| 41行: | 41行: | ||
*スクリーンショット | *スクリーンショット | ||
[[ファイル:Screen.png]] | [[ファイル:Screen.png]] | ||
| + | *課題 | ||
| + | **注目遺伝子があるゲノムが一番上に来ない | ||
| + | **ドメイン飛んでる問題 | ||
| + | **スタンザ化を目指す | ||
== d3sparql.js == | == d3sparql.js == | ||
2015年2月6日 (金) 07:29時点における最新版
目次 |
MBGD
データの準備 → BH14.14/MBGD
シンテニーの可視化
SPARQL
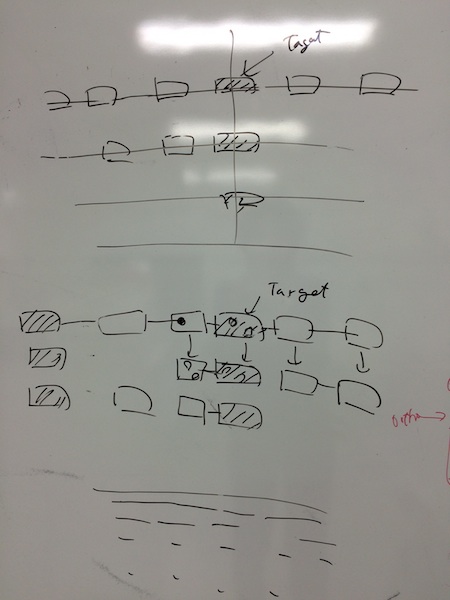
- スタートはMBGD遺伝子名(この例では eco:B2934、セットを小さくするためにセットtax562を指定)
- gene: 中央に置かれる遺伝子
- neibour: その上流下流3000bp内にある遺伝子
- 含まれるオーソログクラスタが二種類以上ある場合は、それぞれ図を描きたい
- MBGD SPARQLポータルサイト: http://mbgd.genome.ad.jp/sparql
PREFIX orth: <http://purl.jp/bio/11/orth#> PREFIX org: <http://mbgd.genome.ad.jp/rdf/resource/organism/> PREFIX faldo: <http://biohackathon.org/resource/faldo#> PREFIX mbgdr: <http://mbgd.genome.ad.jp/rdf/resource/> PREFIX mbgd: <http://purl.jp/bio/11/mbgd#> SELECT DISTINCT ?organism ?group ?gene ?neibour ?genebegin ?nbegin WHERE { ?group orth:inDataset mbgdr:tax562. ?group orth:member/orth:gene <http://mbgd.genome.ad.jp/rdf/resource/gene/eco:B2934>. ?group orth:member/orth:gene ?gene. ?gene orth:organism ?organism. ?neibour a mbgd:CDS. ?neibour orth:organism ?organism. ?gene faldo:location/faldo:begin/faldo:position ?genebegin. ?gene faldo:location/faldo:end/faldo:position ?geneend. ?neibour faldo:location/faldo:end/faldo:position ?nend. ?neibour faldo:location/faldo:begin/faldo:position ?nbegin. FILTER (?genebegin - 3000 <= ?nbegin && ?nend <= ?geneend + 3000) }
Visualize
- 一遺伝子一件の連想配列で戻ったJSONデータを、D3が使いやすい構造に変換
- 同じグループにある遺伝子を同じ色に設定
- D3で描画
- スクリーンショット
- 課題
- 注目遺伝子があるゲノムが一番上に来ない
- ドメイン飛んでる問題
- スタンザ化を目指す
d3sparql.js
- https://github.com/ktym/d3sparql
- README.md を記述 (2015/2/3)
- 可視化を差し込む場所を指定するパッチ取り込み (2015/2/3)
- 可視化を差し込む場所をカスタマイズ可能に改良 (2015/2/4)
- 可視化を差し込む前に以前の可視化を消去するように改良 → 可視化が追加されていくのではなく更新可能に (2015/2/6)
- ファセット
- アプリ開発
WebGL
- WebGLを用いた可視化のサンプル
- 可視化のサンプル(Chrome推奨)
- 作者は,大東誠(makoto.ohigashi@etude.ocn.ne.jp)氏
- 内部で,three.js を使用
- スタンザ