高速アミノ酸配列検索
提供:TogoWiki
目次 |
概要
- アミノ酸配列の検索に特化したサイト
- 短いアミノ酸配列をミスマッチを許容しつつ高速に検索したい
- 高速ゲノム検索GGGenomeのアミノ酸配列版に相当
- MS解析などに役立つ
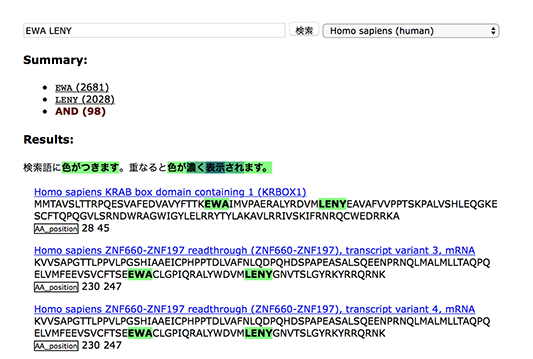
- サンプル画像
検索アルゴリズムとインターフェース
- 検索キーワードはアミノ酸配列のみ
- 最短3残基から検索可能
- ヒットのposition(先頭からの残基数)がわかるようにする
- ミスマッチを許容して検索
- アミノ酸置換マトリクスは使用せず、ミスマッチ残基数のみ考慮
- クエリの例
- ACDEW または 0:ACDEW → 完全一致検索
- 1:ACDEW → 1ミスマッチまで許容
- 2:ACDEW → 2ミスマッチまで許容、以下同様
- 複数のアミノ酸配列のAND検索、OR検索に対応
- 検索語ごとにヒット件数を表示。例:
- FCW (23)
- 1:ACDE (125)
- AND (3)
- OR検索のヒット件数は、個々のクエリのヒット件数を足したものに概ね一致するので、わざわざ正確に計算して表示しなくてもよい
- OR検索の結果一覧(ヒットをハイライト表示)がほしい
- 複数のモチーフがどのようにヒットしているか眺めたい
- 重なる部分は濃く表示、ゆくゆくはキーワードごとに色分け
- 大きなDBを分割しOR検索で分散化できると良い。
- たとえばDADをDIVISIONごとにインデックスし、OR検索によってDAD全体を検索。
- 検索語ごとにヒット件数を表示。例:
- 検索結果のランキング
- ヒットしたキーワードの種類が多い順
- 同じ場合は、ミスマッチの数が少ない順
- REST APIを提供
- HTML / TSV / CSV / JSON で出力
- http://◯◯/uniprot/FCW+1:ACDE.csv のような感じ
- デフォルトはAND検索、URL内では “+" で連結
- ORまたは | をはさむとOR検索、URL内では "|" (%7C) で連結?
検索したいアミノ酸配列のデータベース
アミノ酸配列のmulti-FASTAを作れば簡単にDBを追加できるようにしたい。
→ FASTAを用意すればほぼ自動でindexを作製できるようにした(2017-09-13)
- UniProt
- UniProt Downloads - http://www.uniprot.org/downloads
- Reviewed (Swiss-Prot) - アノテーション済
- Release 2017_08 配列数: 555,426 残基数: 198,919,479 (189.7MiB)
- Unreviewed (TrEMBL) - EMBLを翻訳しただけ
- Release 2017_08 配列数: 89,396,316 残基数: 30,109,048,623 (28.0GiB)
- 上記の2つを合わせたものが、UniProt全体
- まずはヒトのsubsetを対象とする
- seqkit grep -nrp '_HUMAN' でOK
- Reviewed (Swiss-Prot) → 配列数: 20,218 残基数: 11,330,825 (10.8MiB)
- Unreviewed (TrEMBL) → 配列数: 140,037 残基数: 38,772,049 (37.0MiB)
- または、Reference proteomes のリンクから下記のファイルを取得する
- UP000005640_9606.fasta.gz 配列数: 20,984 残基数: 11,484,514 (11.0MiB)
- UP000005640_9606_additional.fasta.gz 配列数: 72,604 残基数: 25,634,242 (24.4MiB)
- seqkit grep -nrp '_HUMAN' でOK
- RefSeqのタンパク (NP_* / XP_*)
- Release 83 (2017/7) 配列数: 88,385,530 残基数: 34,113,050,666 (31.8GiB)
- FASTA: ftp://ftp.ncbi.nlm.nih.gov/refseq/release/complete/ → complete.*.protein.faa.gz
- まずはヒトのsubsetを対象とする
- seqkit grep -nrp '\[Homo sapiens\]' でOK
- GENCODEのタンパク
- RefSeqよりもtranscriptのvariantが網羅されている
- ヒト Release 27 (2017/1) 配列数: 95,659 残基数: 35,656,600 (34.0MiB)
- FASTA: ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_27/gencode.v27.pc_translations.fa.gz
- (ヒト)ゲノム
- 全フレーム翻訳してアミノ酸配列に変換したもの(〜6G残基)を検索
- 未知のペプチドをゲノムから探せるようにする
- tblastnのような感じ
- ヒットした場合、ゲノムの座標を逆算して表示。既知のタンパクかどうかわかるとなお良い。
- 疾患ゲノム(リファレンスゲノムとの差分だけで十分)
- 疾患固有のタンパクを検索できるようにする
- リファレンスゲノムとの差分だけを集めたdecoy配列を生成する方法。ToMMoに相談してみる。
- 全フレーム翻訳してアミノ酸配列に変換したもの(〜6G残基)を検索
- DAD
- DDBJ (trad) に含まれるtranslationを集めたもの。tradと同じDIVに分かれている。
- Release 79.0 (2017/6) 配列数: 63,670,485 残基数: 19,963,617,826 (18.6GiB)
- 1つの塩基配列エントリに複数のCDSが含まれる場合は、個別にIDが付与されている。
- U43141.1 | Human immunodeficiency virus type 1 complete genome
- U43141-1 | AAA85230.1 | Human immunodeficiency virus 1 gag protein
- U43141-2 | AAA85231.1 | Human immunodeficiency virus 1 pol protein
- PDB
- アミノ酸配列から立体構造を検索
- Snapshot 20170710 エントリ数: 131,667 配列数: 408,865 残基数: 101,431,745 (96.7MiB)
- FASTA: ftp://snapshots.pdbj.org/20170710/pub/pdb/derived_data/pdb_seqres.txt.gz (解凍後121MB)
検討事項
- サービスの名称
- GGGenome(ゲゲゲノム)様のネーミングを踏襲しPPProt(プププロット)、PPPeptide(ぺぺペプチド)などが候補
- PPとは?(ねたがみつからない)
- GGGenome(ゲゲゲノム)様のネーミングを踏襲しPPProt(プププロット)、PPPeptide(ぺぺペプチド)などが候補