SPARQLthon24
提供:TogoWiki
第24回 SPARQLthon を下記日程で開催したいと思います。
目次 |
開催概要
- 開催期間:2014年9月25日(木) 10:00 〜 26日(金) 18:00
- 開催場所:ライフサイエンス統合データベースセンター (DBCLS) @ 東京大学 柏の葉キャンパス駅前 サテライト 6階
- アクセス:http://dbcls.rois.ac.jp/access
- 開催連絡:http://groups.google.com/group/biohackathon-jp メーリングリストにて
プロジェクト
TPP グループ全体
- 生物種メタボロームモデル・データベースの構築 (有田)
- D2RQ を使った SPARQL 検索(櫻井)
- → サーバを立ち上げて SPARQL が叩けるようになった。今後マッピングの検討を行う。
- ゲノム・メタゲノム情報統合による微生物DBの超高度化推進 (黒川)
- オーソログと感染症の関係のスタンザの続き (森、千葉、山本)
- GenomeRefine + MicrobeDB.jp の連携(森)
- ユーザのゲノム・メタゲノムデータを GenomeRefine から MicrobeDB.jp に入れる
- FASTA, FASTQ から MeGap, MiGAP で TSV を生成し GenomeRefine が RDF を出力して MicrobeDB.jp へ
- → データをどのように交換するかのフローについて決定した
- 疾患ヒトゲノム変異の生物学的機能注釈を目指した多階層オミクスデータの統合 (菅野)
- 変異などのサンプルデータをもらったので MySQL に入れて、[D2RQ] (内部のみ) で SPARQL 検索できるように(河野)
- まずはデータの中身を調べることから(テーブル数は 80 くらい、染色体ごとに分かれているなど、スキーマが何パターンあるか)
- 既存オントロジーのマッピングを先に行うか、作成する TogoStanza/アプリケーション を考えながらオントロジーを設計するか
- → D2RQ で SPARQL エンドポイントを構築した
- 変異などのサンプルデータをもらったので MySQL に入れて、[D2RQ] (内部のみ) で SPARQL 検索できるように(河野)
- 植物ゲノム情報活用のための統合研究基盤の構築 (田畑)
- 植物学会の辞書(日・英)などから TPP で作成するオントロジーと Plant オントロジー(英・日・西)との関連づけを進める(市原)
- 辞書ごとの語彙の違いのマッピングは手作業でやらざるを得ない(コンフリクトの解消は政治的)
- 何のため(ファセット検索など)のためにオントロジーを作るといった目的を決めるか、語彙全体のカバレッジを上げることを目指すのか
- 植物学会の辞書(日・英)などから TPP で作成するオントロジーと Plant オントロジー(英・日・西)との関連づけを進める(市原)
- 糖鎖統合データベースおよび国際糖鎖構造リポジトリの開発 (成松)
- GlycoProt DB のオントロジーを開発する(新町, 鈴木)
- → 8割方完成、オントロジーの可視化ツールが欲しい
- ペプチドの特定の部位に Glycan がのっているデータについてのオントロジー開発(鹿内)
- → 作成中
- レクチンのデータベース LfDB のオントロジー開発(木下)
- → RDF 化を進めているが、一部データプロバイダと相談する必要あり
- GlycoProt DB のオントロジーを開発する(新町, 鈴木)
- 生命と環境のフェノーム統合データベース (桝屋)
- フェノタイプの RDF 化について、他グループとのすりあわせ
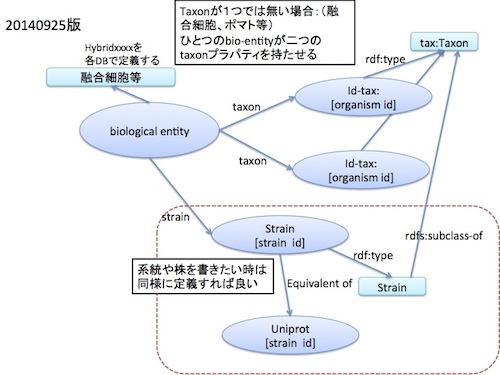
- Biological entityについて: 各回のまとめ
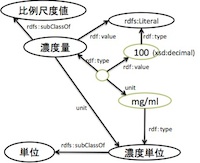
- 数値/定量値について
[ a:濃度量; :value “100”^^xsd:demical; :unit <mg/ml> ]
SPARQLthon グループ
- DDBJ オントロジーの開発
- http://ddbj.nig.ac.jp/ontologies/ で公開予定 (ddbj → tga)
- Taxonomyは毎日、INSDC/DDBJは年1回の更新
- TogoGenome の RDF 更新
- 表現系のデータ更新(川島)
- DDBJ/INSDC オントロジーの最終版にあわせてエントリのメタデータ部分の RDF 化を完成(片山・藤沢)
- 真核ゲノム RDF 化と TogoStanza 開発(守屋・川島・片山)
- 化合物(小寺・山田・時松・櫻井)
- KNApSAcK や日化辞などの DB に含まれている天然化合物を RDF で表現し分類、マスタグの意味付けなどに利用
- 多数の化合物が得られた時に、その化合物を一個一個チェックして共通点を発見するのは辛い。それを自動化する仕組みが欲しい。
- ChEBI のオントロジーが天然物用にはしっくりこないので、オントロジーの整備から
- KEGGやKNApSAcKの化合物を網羅的に眺めて楽しむページ
- JSTの日本化学物質辞書(日化辞)のRDFデータがCC BYで公開される見込み(櫛田)
- TPPの成果DBやアーカイブの収録されているDBの化合物情報を日化辞をハブとして統合することを目指す。
- CHEMINFおよびSIOを使って標準化を進める。
- 参考資料(https://wg.biosciencedbc.jp/lib/exe/fetch.php/nbdc_nikkaji_rdf_20140926.pdf)
- KNApSAcK や日化辞などの DB に含まれている天然化合物を RDF で表現し分類、マスタグの意味付けなどに利用
- がんゲノムの RDF 調査、Linked ICGC (山中)
- ICGC のテーブルからオントロジーへの対応
- SPARQL Builder の改良(小林・レンツ・古崎・呉・山口)
- 改良版 [1] パスのランキング機能,ラベル表示機能,パス数表示機能等追加
- Linked Data 用のオントロジー (VoID) + SPARQL 1.1 用のオントロジー (Service Description) による SPARQL endpoint のメタデータ記述について標準化をすすめたい
- SPARQLTester - エンドポイントテスト用のスクリプトを公開(山本)
- → SPARQLbin と Lodestar をローカルでテストすることに成功 (SPARQLthon/SPARQL_b_i_n, SPARQLthon/LODEStar)
- TripleDataProfilerを用いたSPARQLエンドポイントのメタデータを収集したサイトの公開 [6]
BioHackathon
参考リンク
- これまでの SPARQLthon
参加者
- 片山俊明(DBCLS)
- 川島秀一(DBCLS)
- 大田達郎(DBCLS)
- 小林紀郎 (理研)
- 山口敦子 (DBCLS)
- 藤澤貴智(遺伝研)
- 戀津魁 (理研)
- 岡別府陽子(MSS)
- 山中遼太(先端研)
- 永野朗夫(PENQE)25日のみ
- 千葉啓和(基生研)
- 山田一作(野口研)
- 小寺正明(東工大)
- 山本泰智(DBCLS)
- 森宙史(東工大)25日のみ
- 山本希(東工大)26日のみ
- 守屋勇樹(DBCLS)
- 時松敏明(DBCLS)
- 桝屋啓志(理研BRC)25日のみ
- 高月照江(理研BRC)
- 市原寿子(かずさDNA研)
- 木下聖子(創価大)25日のみ
- 新町大輔(創価大)
- 青木信行(創価大)
- 櫻井望(かずさDNA研)25日のみ
- 西出浩世(基生研)
- 鈴木真也(東工大)25日のみ
- 古崎晃司(大阪大)26日のみ
- 櫛田達矢(NBDC)26日のみ
- 畠中秀樹(NBDC)26日のみ
- 加藤文彦 (ROIS) 25日のみ