SPARQLthon18/Disease
提供:TogoWiki
(版間での差分)
(→辞書的なOWLを使って,ハブ的な使い方をする) |
(→辞書的なOWLを使って,ハブ的な使い方をする) |
||
| 113行: | 113行: | ||
== 辞書的なOWLを使って,ハブ的な使い方をする == | == 辞書的なOWLを使って,ハブ的な使い方をする == | ||
| - | * ICD10の階層を使用して,関連するIDや同義語をマッピングしていく。 | + | * 目的:網羅的な疾患分類と,他のIDと同義語を関連付ける辞書且つハブ的なRDFを作って繋げる。 |
| + | ** ICD10の階層を使用して,関連するIDや同義語をマッピングしていく。 | ||
| + | * ICD10の小分類を1つのIDに対応させ,最も細かい分類,もしくはその上の分類で該当する他のIDや,同義語をマッピングしていく。 | ||
| + | ** ↓叩き台です。叩いてください。 | ||
[[ファイル:icd10mapping.png]] | [[ファイル:icd10mapping.png]] | ||
| + | * 懸念事項。ICD10の変更点も結構多い。←本当にICD10で良いのか。 | ||
| + | * URIをどうするのか。 | ||
=== オントロジー === | === オントロジー === | ||
* Disease Ontology | * Disease Ontology | ||
2014年3月14日 (金) 10:06時点における最新版
目次 |
疾患関連のLinked Data
概要
基礎研究から応用研究まで疾患名がついているデータは数多くあります。ただ,これらの疾患名は現状文字列のみで書かれていたり,揺れがあったり, RDFにおいては,特定の分野のみで使用が可能な,predicateがつけられていたりしていざ疾患名をキーとしてクエリを投げる時に戸惑うことがあります。 icd10やOMIMなど既存のID体系もありますが,疾患の専門家でない人がどうIDを付与すべきかということを考えるのも難しく,疾患分類体系自体も 時間の経過とともにダイナミックに変化してしまうという問題があります。 ここでは,RDF化したデータに疾患名が含まれていた時に,それらをどう扱い,繋げていくのか 疾患をキーとしてクエリを投げたい時に,どのようなクエリが生成されたら良いのかといったことを考えていきたいと思います。
既存のアプローチ
- 当てはまりそうなものをまず探す。
- SPARQL Endpointからデータを取って来て眺める。
- BioPortalのSPARQL endpointを活用して(例,ICD10の場合)
BioPortalのSparql Endpointにアクセス SPARQLのクエリを投げて,その結果から疾患の記述方法や,使えそうなpredicateが無いか探す。
PREFIX omv: <http://omv.ontoware.org/2005/05/ontology#>
SELECT ?s ?p ?o
WHERE {
GRAPH <http://bioportal.bioontology.org/ontologies/ICD10> {
?s ?p ?o.
}
}
LIMIT 100
- 結果
{"head":{"vars":["s","p","o"]},
"results": {
"bindings":[
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.0"},
"p":{"type":"uri","value":"http://www.w3.org/2004/02/skos/core#notation"},
"o":{"type":"literal","value":"N88.0","datatype":"http://www.w3.org/2001/XMLSchema#string"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.0"},
"p":{"type":"uri","value":"http://www.w3.org/2000/01/rdf-schema#subClassOf"},
"o":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.0"},
"p":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/cui"},
"o":{"type":"literal","value":"C0269194","datatype":"http://www.w3.org/2001/XMLSchema#string"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.0"},
"p":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/tui"},
"o":{"type":"literal","value":"T191","datatype":"http://www.w3.org/2001/XMLSchema#string"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.0"},
"p":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/hasSTY"},
"o":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/sty/T191"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.9"},
"p":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/cui"},
"o":{"type":"literal","value":"C0156377","datatype":"http://www.w3.org/2001/XMLSchema#string"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.9"},
"p":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/tui"},
"o":{"type":"literal","value":"T047","datatype":"http://www.w3.org/2001/XMLSchema#string"}},
{"s":{"type":"uri","value":"http://purl.bioontology.org/ontology/ICD10/N88.9"},
"p":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/hasSTY"},
"o":{"type":"uri","value":"http://bioportal.bioontology.org/ontologies/umls/sty/T047"}},
- 汎用的なPredicateを探す
- 既存のものでちょうどよいものが見つからなかった場合,オントロジーごと作成する。
手持ちのデータに疾患名があった場合に”疾患である”ということだけを表記したい場合
アプローチ1
- IDを付与する
- メリット
- 綺麗に疾患間を繋げられる。
- 表記ゆれが無い。
- デメリット
- 付与するのが面倒である。
- 疾患体系が今後変更される可能性がある。
- 付与したIDに責任が持てない。
- メリット
(↑特に実験系の研究者の方にお願いをしようとすると,IDの付与に責任が持てないので外部に公開したくないというようなお返事を頂くことが多いです。)
@prefix db: <http://dbpedia.org/ontology/> . <http://sample.jp/orphan#SampleDisease> --中略-- db:icd10 "E752".
- ↑ここでは,dbpediaを使ってicd10を書いてしまいましたが,他にもっと良い語彙があったら教えて下さい。
アプローチ2
- 疾患名であることのみを示して,疾患名とIDが対応付けられたRDFセット(辞書的なもの)を用いて対応付けられるようにする。
- メリット
- RDF化が簡単。IDをつけたことに対する責任を問われることが無い。
- ID体系が変化しても(2015年にICD11が導入予定)影響は無い。
- デメリット
- 表記ゆれを解消しきることが難しいかもしれない。(データ量による?)
- 生物資源(実験動物)のデータの例(Schema.orgのMedicalConditionを使用)
- メリット
@prefix sample_animalbank: <http://sample.jp/animalbankVocabulary#> .
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
@prefix BioLOD_property_pria315s13i: <http://purl.jp/bio/13/property/pria315s13i/> .
@prefix Medical_condition: <http://schema.org/MedicalCondition/> .
<http://sample.jp/resource/animalbank/nbio155>
sample_animalbank:ID
"sample155";
rdf:type BioLOD_class_cria315s1i:Mouse_Strain;
BioLOD_property_pria315s13i:taxon
<http://purl.jp/bio/13/10090/crib166u26rib166u10090i>;
--中略--
Medical_condition:name "Infection","Immune abnormality".
アプローチ3
- 両者併用
- IDを付与できる場合は付与し,難しい場合は,疾患名のみを記述する。別に作成した辞書的なRDFで両者を繋げられるようにする。
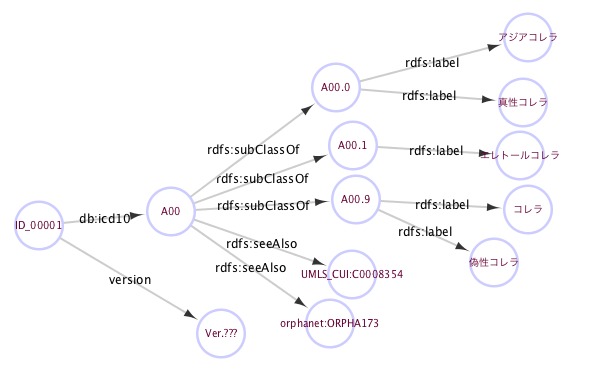
辞書的なOWLを使って,ハブ的な使い方をする
- 目的:網羅的な疾患分類と,他のIDと同義語を関連付ける辞書且つハブ的なRDFを作って繋げる。
- ICD10の階層を使用して,関連するIDや同義語をマッピングしていく。
- ICD10の小分類を1つのIDに対応させ,最も細かい分類,もしくはその上の分類で該当する他のIDや,同義語をマッピングしていく。
- ↓叩き台です。叩いてください。
- 懸念事項。ICD10の変更点も結構多い。←本当にICD10で良いのか。
- URIをどうするのか。
オントロジー
- Disease Ontology
- OBO Foundry
- ICD-10(International Statistical Classification of Diseases and Related Health Problems--国際疾患分類)
- Schema.org Medical Condition
- NCIT (National Cancer Institute Thesaurus)
- MESH (Medical Subject Headings)
- MedDRA (Medical Dictionary for Regulatory Activities) 英語
- MedDRA 日本語
メンバー
伊藤真和吏(NIBIO)