BH13.13/TaxID2BioSampleID
提供:TogoWiki
目次 |
概要
NCBI Taxonomy は、主としてNCBIの塩基配列データベースに登録されている遺伝子が由来する生物種に関して、系統分類と学名に関する情報が収集されたデータベースである。系統分類の各階層にはTaxonomy ID が振られている。これまでは(特に原核生物について問題になることだが)種より細かい系統分類(Strain や Substrain 等)に対してもTaxonomy ID がふられてきた。例えば、大腸菌O157 Sakai株 (Escherichia coli O157:H7 str. Sakai) の場合、ゲノム配列が登録されたSakai株のTaxonomy ID は 386585、一つ上の階層にあたるEscherichia coli O157:H7 は 83334、さらに一つ上の階層にあたるEscherichia coli は 562 というTaxonomy ID がふられている。ランクに関しては、Escherichia coliには、Rank: species と記載されているが、それ以下の階層は全て、Rank: no rank とされている。さて先日、NCBI から、2014 年1月以降、種レベル未満のランクについて Taxonomy ID の新規発行をやらなくなる、というアナウンスがあった。詳細は、参考リンクや以下に譲るが、今後はTaxonomy ID に変わって、BioSample ID が、配列情報が由来する生物をユニークに区別するIDとして利用されていくことになる。
これまで、TogoGenome 等のシステムでは、生物種がRDFの主語になる場合にTaxonomy ID を利用してきたが、新規にゲノムが決まった生物サンプルはTaxonomy IDを持たないことから、主語としてもBioSample IDを利用していくことになると考えられる。配列情報から生物種へリンクするためのIDとしてTaxonomy IDは、広く利用されてきたこともあり、今回のNCBIの決定は、他のRDF構築にも影響すると考えられる(例えば、菌株保存センターのRDF等)。本グループでは、各種ライフサイエンス関連RDFのリンク関係が今後も破綻なく維持されることを目標に、Taxonomy ID から Sample ID への移行が引き起こす可能性のある、RDF構築上の問題点について洗い出して今後の開発方針を決め、RDF/オントロジー開発者間の意識共有を行う。
想定される問題
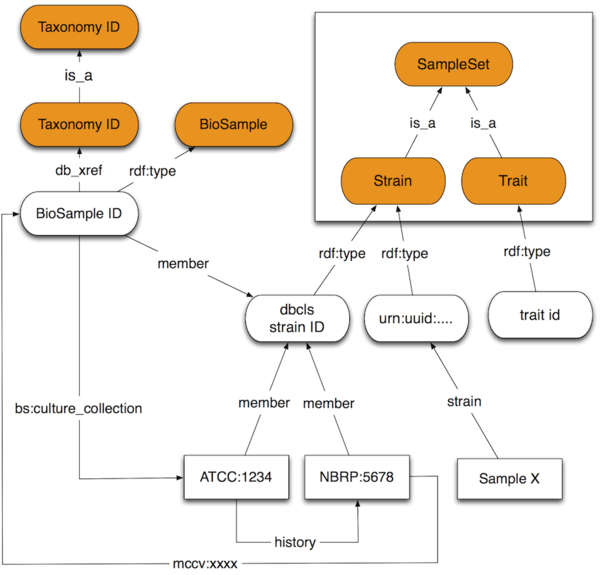
菌株保存センター由来のサンプル
すでに、ATCC株についてはNCBIによってユニークなBioSample IDが発行されている。今後、例えばDDBJにより日本の菌株保存センターの菌株にユニークなBioSample IDが発行された場合、来歴上同じ菌株とされている菌株に異なるBioSample IDが存在することになる。例えば、ゲノムを決めたのがATCC株だった場合、TogoGenome のRDFの主語は、そのATCC株のBioSample IDになるが、来歴上同じ菌株とされているNBRC株のメタデータを利用したい場合、これまでのようにTaxonomy IDを利用した統合はできず、来歴を調べて統合することになる。
ATCC株のBioSampleエントリーの例 http://www.ncbi.nlm.nih.gov/biosample/2472057
1/27
- 統合プロジェクト連携でサンプル情報から生物種の情報をtaxonomy IDで統合する提案
- Strain IDレベルのTaxonomy IDを今後ふらない問題を共有
- 対応案と生物種による統合に向けてBioSample IDを持たない(INSDCに登録のない)サンプル情報についても表現できるモデル概要を設計
- Taxonomy IDのサブクラスとしてStrain, Cultivarを指し示すTaxonomic Subtypeクラス作成し、スモールオントロジーを開発の合意
1/28
- 1/27でデザインしたモデル概要を踏まえてMicrobeDB.jp, DBCLSのメンバーにてクラス, インスタンスの確認しながら詳細RDFデータモデルを設計
- DDBJメンバーによるBioSample ID間の関係を記述するtypeRelationのxsd定義の確認と現在のNCBI BioSampleデータのRDF化の準備