BH13.13/Arabidopsis
提供:TogoWiki
(→背景) |
(→背景) |
||
| 26行: | 26行: | ||
* 全databaseのontology情報の活用 | * 全databaseのontology情報の活用 | ||
** model中に含まれるreactionに対応するPIERO ontologyを利用した解析を行う | ** model中に含まれるreactionに対応するPIERO ontologyを利用した解析を行う | ||
| + | ** KNApSAcK のmetadataを利用した解析を行う | ||
** KEGGのpathway layoutを活用した可視化 | ** KEGGのpathway layoutを活用した可視化 | ||
2014年1月31日 (金) 03:23時点における最新版
目次 |
参加者
福島(理研CSRS)、西田(理研QBiC)
目的
genome scale metabolic model, AraCyc, KEGG, KNApSAcK 全てのlinked data化と、このlinked dataとomics dataを利用したapplicationの公開。
(上記実現に向けたdata調査、pathway 作成とそのturtle fileの試作を行う。)
背景
Arabidopsis の metabolic model reconstruction としては下記のようなものがある。
- Poolman's model(http://www.ncbi.nlm.nih.gov/pubmed/19755544) 2009年
- AraGEM(http://www.ncbi.nlm.nih.gov/pubmed/20044452) 2010年
- Mintz-Oron's model(http://www.ncbi.nlm.nih.gov/pubmed/22184215) 2012年
これらのmodelはFBA等を用いてgenomeから代謝反応までの関連付けを可能な限り増やしたものであるが、
modelをomics解析に利用する際に下記が困難という問題がある。
- model, AraCyc, KEGG, KNApSAcK 間の差分の確認
- tableのdump、KEGGのようなpathway diagram上でのhighlight
- script言語を用いたdataのparse、join、また手作業でのmergeを必要としないdata統合
- 全databaseのontology情報の活用
- model中に含まれるreactionに対応するPIERO ontologyを利用した解析を行う
- KNApSAcK のmetadataを利用した解析を行う
- KEGGのpathway layoutを活用した可視化
その理由として各dataのopen linked data化がまだ未熟であることが挙げられる。
(ただし、AraCycについてはBioLODからRDF fileが入手可能。 https://biolod.org/database/rib39i/AraCyc_Pathway_BioPAX_Level2)
手法
そこで本groupではflavonoid pathwayの一部にしぼったTurtle fileとこれに対するSPARQL queryの試作を行った。
- reactionのontologyには小寺さんからご提供いただいたPIERO ontology turtle fileを用いた
- AGI(Arabidopsis Genome Initiative)のgene codeが付けられたmicroarray profileとmass profileを統合解析するuse caseを想定し、AGI codeからKNApSAcKのdataまでのlinkを確認するusecaseを想定
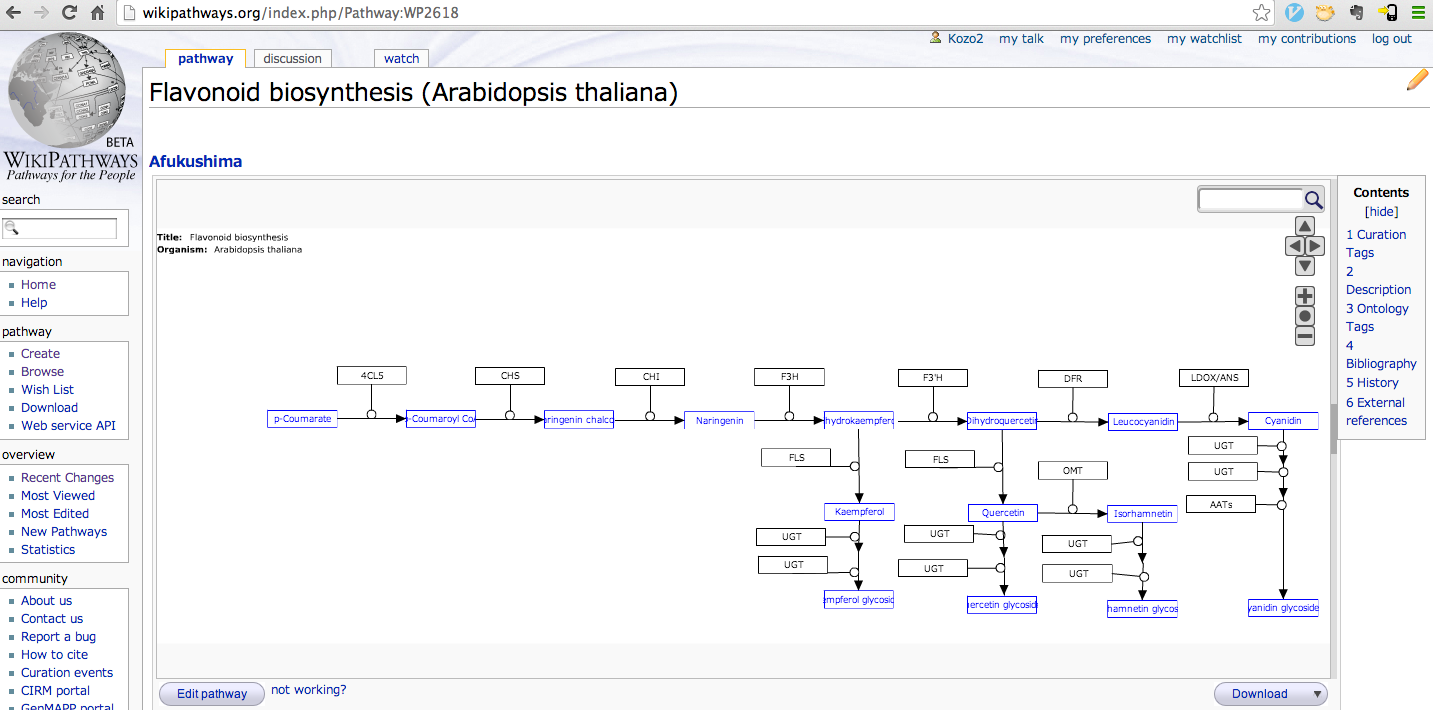
対象としたflavonoid pathway中のgene productとreactionの関係はwikipathways http://wikipathways.org/index.php/Pathway:WP2618 から確認できる
作成したTurtle fileは下記
- predicate piero:catalyzes の subject は本来はEnzymeだが頂いたversionでのPIERO ontologyにはまだ piero:encodes predicateが無かったためsubjectをEnzyme gene(uniprotのURI)としている。
@prefix knapsack: <http://kanaya.naist.jp/knapsack_jsp/information.jsp?sname=C_ID&word=> . <http://purl.uniprot.org/tair/AT5G13930> piero:catalyzes kegg:R01613 . <http://purl.uniprot.org/tair/AT3G55120> piero:catalyzes kegg:R02446 . <http://purl.uniprot.org/tair/AT5G42800> piero:catalyzes kegg:R03636 . <http://purl.uniprot.org/tair/AT4G22880> piero:catalyzes kegg:R05036 . <http://purl.uniprot.org/tair/AT5G08640> piero:catalyzes kegg:R03126 . kegg:R01613 piero:hasReactant kegg:C00223 . kegg:R01613 piero:hasReactant kegg:C06561 . kegg:R02446 piero:hasReactant kegg:C00509 . kegg:R02446 piero:hasReactant kegg:C06561 . kegg:R03636 piero:hasReactant kegg:C05906 . kegg:R03636 piero:hasReactant kegg:C01617 . kegg:R05036 piero:hasReactant kegg:C05905 . kegg:R05036 piero:hasReactant kegg:C05906 . kegg:R03126 piero:hasReactant kegg:C00974 . kegg:R03126 piero:hasReactant kegg:C05903 . kegg:C06561 rdfs:seeAlso knapsack:C00007233 . kegg:C00509 rdfs:seeAlso knapsack:C00000982 . kegg:C05906 rdfs:seeAlso knapsack:C00008991 . kegg:C01617 rdfs:seeAlso knapsack:C00000677 . kegg:C05905 rdfs:seeAlso knapsack:C00006614 . kegg:C00974 rdfs:seeAlso knapsack:C00007234 . kegg:C05903 rdfs:seeAlso knapsack:C00004565 .
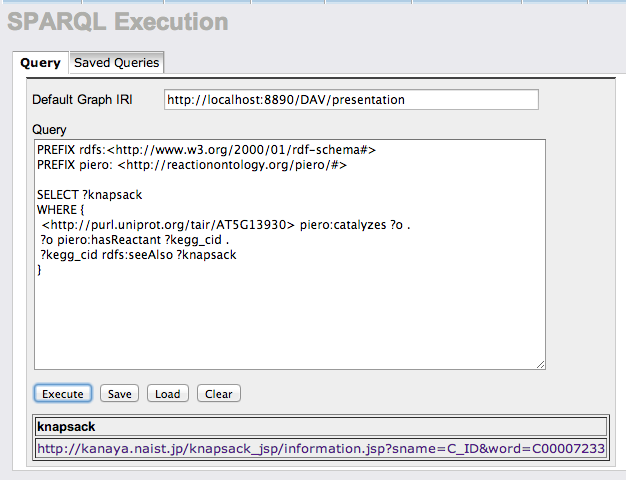
上記Turtleと小寺さんのPIERO Turtle fileをmergeしたものをvirtuosoにimportし下記のSPARQLを適用した。
PREFIX rdfs:<http://www.w3.org/2000/01/rdf-schema#>
PREFIX piero: <http://reactionontology.org/piero/#>
SELECT ?knapsack
WHERE {
<http://purl.uniprot.org/tair/AT5G13930> piero:catalyzes ?o .
?o piero:hasReactant ?kegg_cid .
?kegg_cid rdfs:seeAlso ?knapsack
}
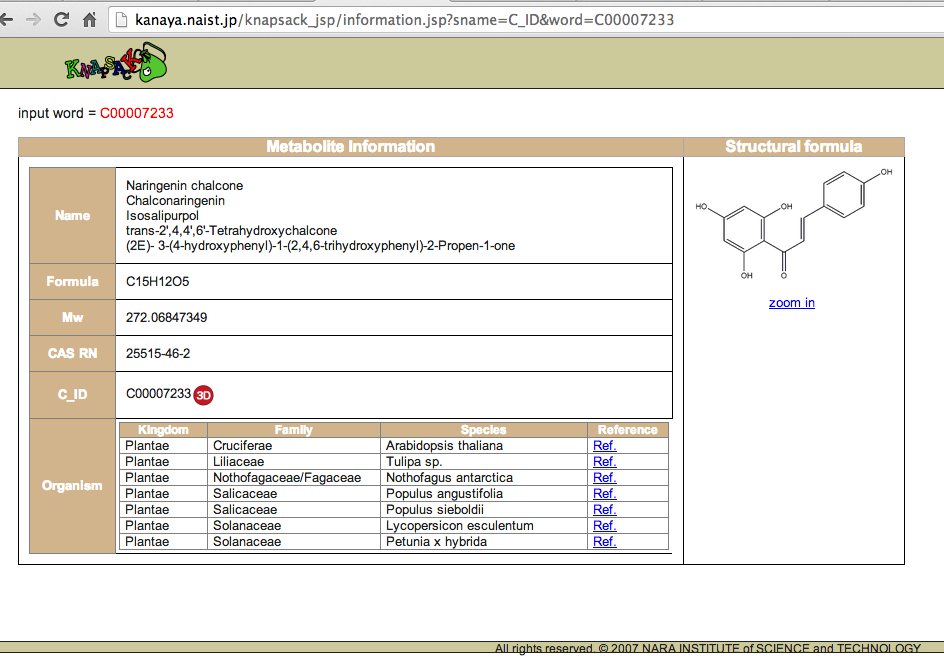
これによりAGI code AT5G13930に対応付けられたKNApSAcK中のcompound C00007233 へのlinkを容易に得ることが可能となった。
結果と考察
TAIR, KEGG, KNApSAcKをつなぐRDFの1例を示した。 RDF導入により
- 差分確認 (http://www.genome.jp/dbget-bin/www_bget?C00223 からKNApSAcKへのlinkは無いこと)
- 異種(遺伝子、化合物) entry間の対応付け
が統一的にあつかえることが明らかとなった。
このように遺伝子、化合物の対応付けが容易になることで発現、mass profileの統合解析applicationの作成を推進することが期待される。
memo
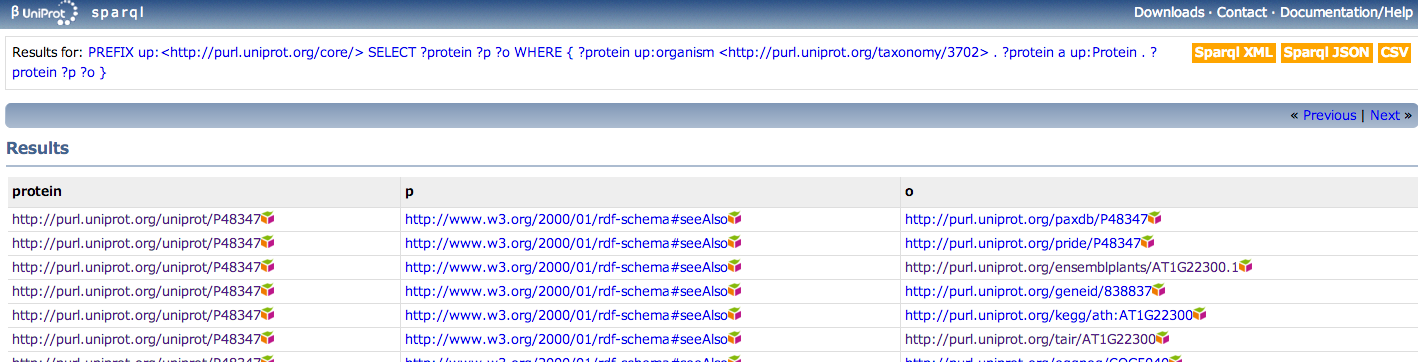
河野さんのuniprot調査結果
省略記法、以降略記up:が利用可能
PREFIX up:<http://purl.uniprot.org/core/>
Arabidopsis(taxonomy id 3702) のprotein entry pageへのURIのlistを得る。 a は rdf:type の省略記法
PREFIX up:<http://purl.uniprot.org/core/>
SELECT ?protein
WHERE
{
?protein up:organism <http://purl.uniprot.org/taxonomy/3702> .
?protein a up:Protein .
} LIMIT 10
Arabiのproteinにどのようなpredicateがあるか調べる。
PREFIX up:<http://purl.uniprot.org/core/>
SELECT DISTINCT ?p
WHERE
{
?protein up:organism <http://purl.uniprot.org/taxonomy/3702> .
?protein a up:Protein .
?protein ?p ?o
}
Arabiのproteinが持っている情報を全て出力する。
PREFIX up:<http://purl.uniprot.org/core/>
SELECT ?protein ?p ?o
WHERE
{
?protein up:organism <http://purl.uniprot.org/taxonomy/3702> .
?protein a up:Protein .
?protein ?p ?o
}
手持ちのdataがTAIRに基づいたものである場合のuniprot rdf探索の手順
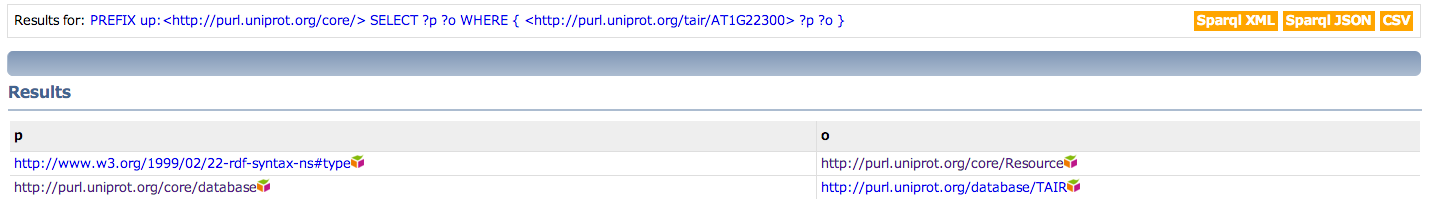
1. まず適当なTAIRのURIからoとするuriを調べる。TAIRのURIは前述のSPARQL結果からわかる。
PREFIX up:<http://purl.uniprot.org/core/>
SELECT ?p ?o
WHERE
{
<http://purl.uniprot.org/tair/AT1G22300> ?p ?o
}
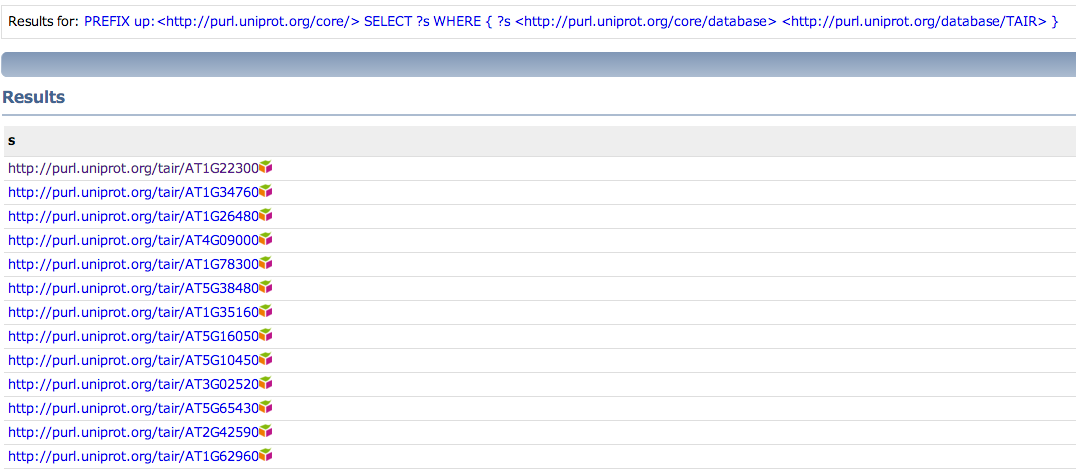
2. 1. からTAIRへのlinkがあるURIのlistは下記で得られる
PREFIX up:<http://purl.uniprot.org/core/>
SELECT ?s
WHERE
{
?s <http://purl.uniprot.org/core/database> <http://purl.uniprot.org/database/TAIR>
}
3. 手持ちのTAIRのATG code AT1G22300に対応するUniprot entry URIを得る
#PREFIX rdfs:<http://www.w3.org/2000/01/rdf-schema#>
SELECT ?s
WHERE
{
?s rdfs:seeAlso <http://purl.uniprot.org/tair/AT1G22300>
}