BH11.11/生物学辞書のRDF化
提供:TogoWiki
DBCLSの生物学辞書 をRDF化する.
目次 |
データについて
ファイルは以下の二つ(+一つ).
- japanese_species_names.utf8.txt (種名)
- japanese_scientific_terms.utf8.txt (科学用語)
- sci_terms.txt (NIIオンライン用語)
元データ形式
@…@ is @…@ for @…@ in 典拠1 典拠2 version バージョン名
O P S
RDF変換
合計トリプル数: 4,070,297 (2011/11/24) 変換時間: species_names (12時間), scientific_terms(1時間) ファイルサイズ: 1GBくらい species_names (TriX 850MB程度), scientific_terms (TriX 108MB)
predicate のマッピング => Google Spreadsheet
scientific_termsはリンクを大分整理したのでトリプル数減るはず.
データモデル
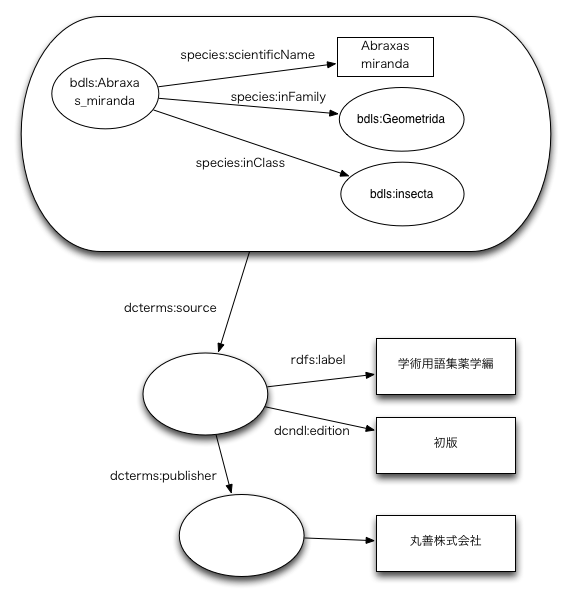
3つ組の由来が何であるかが重要なので,Named Graphで記述するようにする.
S P O G .
G 由来 典拠2 .
典拠2 version バージョン名
データ提供/出版社? 典拠1 .
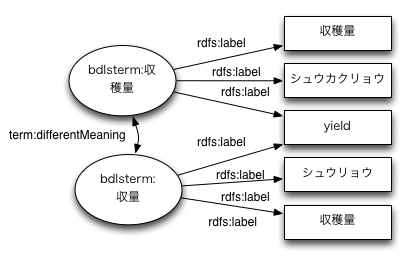
学名-和名間 / 和名-和名間 => skos:closeMatch ?
学術用語は日本語の異義語関係が重要らしい.
URI設計
- Graph/Source URIの振り方を工夫すべき (MySQL?)
S/OのURI
学名については http://lod.ac/bdls/学名 でやってみる.学名のスペースは_に 用語のほうはそのまま使うと記号で苦労しそう. <= 学名も用語も独自でID振ったほうがよいかも
科学用語と生物種のBASE_URIは分ける
科学用語の中に,科についての情報は入っている => 生物種のほうとskos:exactMatch辺りで結んであげるべき?
Graph URI
典拠1+典拠2+バージョンの組み合わせでシリアル
由来の記述
provenance 関係のontologyが参考になるかもしれない.ヘビーという意見もある.
- pav-ontology - PAV 2.0 - Provenance Authoring and Versioning ontology - Google Project Hosting
- The PROV Data Model and Abstract Syntax Notation
TriXの例
とりあえずgraph uri含めて表現できるのでTriXでやっている. RDF.rbのnquadsは\u escape sequenceになる. turtleベースのTriGのほうが好みだけどRDF.rbにライブラリがない. <= 作るべき
<graph>
<triple>
<uri>http://lod.ac/bdls/graph/17</uri>
<uri>http://purl.org/dc/terms/source</uri>
<uri>http://lod.ac/bdls/source/17</uri>
</triple>
<triple>
<uri>http://lod.ac/bdls/source/17</uri>
<uri>http://www.w3.org/2000/01/rdf-schema#label</uri>
<plainLiteral xml:lang="ja">日本産 魚類検索 全種の同定 第二版</plainLiteral>
</triple>
<triple>
<uri>http://lod.ac/bdls/source/17</uri>
<uri>http://ndl.go.jp/dcndl/terms/edition</uri>
<plainLiteral xml:lang="ja">version @2000 年 12 月 20 日 第 1 版第 1 刷@</plainLiteral>
</triple>
</graph>
<graph>
<uri>http://lod.ac/bdls/graph/17</uri>
<triple>
<uri>http://lod.ac/bdls/Abalistes_stellaris</uri>
<uri>http://lod.ac/ns/species#scientificName</uri>
<plainLiteral>Abalistes stellaris</plainLiteral>
</triple>
<triple>
<uri>http://lod.ac/bdls/Abalistes_stellaris</uri>
<uri>http://lod.ac/ns/species#inGenus</uri>
<uri>http://lod.ac/bdls/(オキハギ属)</uri>
</triple>
<triple>
<uri>http://lod.ac/bdls/Abalistes_stellaris</uri>
<uri>http://lod.ac/ns/species#commonName</uri>
<plainLiteral xml:lang="ja">オキハギ</plainLiteral>
</triple>
<triple>
<uri>http://lod.ac/bdls/Abalistes_stellaris</uri>
<uri>http://lod.ac/ns/species#authority</uri>
<plainLiteral>(Bloch and Schneider)</plainLiteral>
</triple>
<triple>
<uri>http://lod.ac/bdls/(オキハギ属)</uri>
<uri>http://www.w3.org/2000/01/rdf-schema#label</uri>
<plainLiteral xml:lang="en">(オキハギ属)</plainLiteral>
</triple>
</graph>
ユースケース
- 生物名辞書から和名“アミシダ”に対応する学名を探すファイル:辞書のRDF化とユースケース.pdf
蝶データからのリンク
以前作った蝶のデータから生物学辞書のRDFに当ててみて,どれくらい当たるか見る.そのためには、双方のデータをマッチングする必要がある。そのような要求を解決するためのツールとしてSilkがある。
- Silkのテスト
Silkが使えるかテストしてみた。 テストは、LODACの博物館情報とLODAC生物情報の標本が収蔵されている博物館情報とのマッチング。
- インストール
- 設定ファイルの作成
- Prefixタグ
- DataSourcesタグ
- LinkTypeタグ
- SourceDatasetタグ・TargetDatasetタグ
- Aggregateタグ・Compareタグ
- Filterタグ
- Outputタグ
- Silkの実行
- 実行結果
Silkから、Silkのバージョン2.5.2をダウンロードして解凍。
test.xmlというファイル名で下記の内容の設定ファイルを作成。設定方法の原文はこちら。
<?xml version="1.0" encoding="utf-8" ?>
<Silk>
<Prefixes>
<Prefix id="rdf" namespace="http://www.w3.org/1999/02/22-rdf-syntax-ns#" />
<Prefix id="rdfs" namespace="http://www.w3.org/2000/01/rdf-schema#" />
<Prefix id="species" namespace="http://lod.ac/ns/species#" />
<Prefix id="owl" namespace="http://www.w3.org/2002/07/owl#" />
<Prefix id="foaf" namespace="http://xmlns.com/foaf/0.1/" />
</Prefixes>
<DataSources>
<DataSource id="lodac" type="sparqlEndpoint">
<Param name="endpointURI" value="http://lod.ac/sparql" />
</DataSource>
<DataSource id="species" type="sparqlEndpoint">
<Param name="endpointURI" value="http://lod.ac/species/sparql" />
</DataSource>
</DataSources>
<Interlinks>
<Interlink id="museumName">
<LinkType>owl:sameAs</LinkType>
<SourceDataset dataSource="lodac" var="a">
<RestrictTo>
?a rdf:type foaf:Organization
</RestrictTo>
</SourceDataset>
<TargetDataset dataSource="species" var="b">
<RestrictTo>
?b species:museumName ?o
</RestrictTo>
</TargetDataset>
<LinkageRule>
<Aggregate type="average">
<Compare metric="levenshteinDistance" threshold="2.0">
<Input path="?a/rdfs:label[@lang='ja']" />
<Input path="?b/species:museumName" />
</Compare>
</Aggregate>
</LinkageRule>
<Filter />
<Outputs>
<Output type="file" minConfidence="0.95">
<Param name="file" value="accepted_links.nt" />
<Param name="format" value="ntriples" />
</Output>
<Output type="file" maxConfidence="0.95">
<Param name="file" value="verify_links.nt" />
<Param name="format" value="alignment" />
</Output>
</Outputs>
</Interlink>
</Interlinks>
</Silk>
使用するnamespaceのprefixを設定する。
マッチング対象のデータソースを指定する。今回のテストでは、LODACで公開したsparql endpointを設定した。idは、SourceDatasetタグで使用するので、データソースを判別しやすい名前をつけておくとよい。
マッチングした結果をリンクするためのプロパティを記述する。
実際に比較するデータを設定する。今回は、DataSourceタグでsparqlEndpointを指定したので、RestrictToタグ内に、sparqlクエリ(のような?形式)を記述する。
マッチング・比較する方法を設定する。Inputタグには、XPath(のような?形式)でマッチング・比較するデータを設定する。
今回は未設定。
出力する閾値とファイル名、フォーマットを指定する。
実行コマンドは、次の通り。
java -DconfigFile=test.xml -jar C:\hogehoge\silk.jar
実行結果は、.silk\outputディレクトリに生成される。今回のテストデータは、LODAC側が約20万件、LODAC生物情報が約120万件なので、20万×120万=2400億回のマッチングを行った計算になるが、実行時間は、開始19:00から終了5:55の約11時間。今回、マッチングの判定にlevenshteinDistanceを使ったが、単純比較なのでequalityを使った方が早かったかもしれない。 accepted_links.nt(ntriples形式)の結果(一部)は次の通り。565615件(標本データの半分弱)ヒットした。
<http://lod.ac/id/524796> <http://www.w3.org/2002/07/owl#sameAs> <http://lod.ac/species/specimen/k59329> . <http://lod.ac/id/524796> <http://www.w3.org/2002/07/owl#sameAs> <http://lod.ac/species/specimen/k59328> . <http://lod.ac/id/524796> <http://www.w3.org/2002/07/owl#sameAs> <http://lod.ac/species/specimen/k59327> . : <http://lod.ac/id/8223> <http://www.w3.org/2002/07/owl#sameAs> <http://lod.ac/species/specimen/k179836> . <http://lod.ac/id/8223> <http://www.w3.org/2002/07/owl#sameAs> <http://lod.ac/species/specimen/k179835> . <http://lod.ac/id/8223> <http://www.w3.org/2002/07/owl#sameAs> <http://lod.ac/species/specimen/k179834> . :
minConfidence属性で、0.95と設定したため、マッチングできた物が返ってきた。 verify_links.nt(alignment形式)の結果(一部)は次の通り。
<?xml version='1.0' encoding='utf-8' standalone='no'?>
<rdf:RDF xmlns='http://knowledgeweb.semanticweb.org/heterogeneity/alignment#'
xmlns:rdf='http://www.w3.org/1999/02/22-rdf-syntax-ns#'
xmlns:xsd='http://www.w3.org/2001/XMLSchema#'
xmlns:align='http://knowledgeweb.semanticweb.org/heterogeneity/alignment#'>
<Alignment>
<map>
<Cell>
<entity1 rdf:resource="http://lod.ac/id/400651"></entity1>
<entity2 rdf:resource="http://lod.ac/species/specimen/k1173634"></entity2>
<relation>
=
</relation>
<measure rdf:datatype="http://www.w3.org/2001/XMLSchema#float">
Some(0.0)
</measure>
</Cell>
</map>
<map>
<Cell>
<entity1 rdf:resource="http://lod.ac/id/400651"></entity1>
<entity2 rdf:resource="http://lod.ac/species/specimen/k1037689"></entity2>
<relation>
=
</relation>
<measure rdf:datatype="http://www.w3.org/2001/XMLSchema#float">
Some(0.0)
</measure>
</Cell>
</map>
:
maxConfidence属性で、0.95と設定したため、マッチングできなかった物が返ってきた。Some(0.0)という記述がマッチングできなかったことを示していると思われる。
- マッチングついでに
標本データにある博物館でLODACに無いものをリストアップしてみた。リストアップには、上記のマッチング結果から、マッチングできなかった標本IDを抽出し、その標本IDに対応する博物館をsparqlで取得する方法を用いた(もっと効率的な方法があったかもしれない)。 結果は、次の通り。
| 博物館名 | 対応するLODACの博物館 | コメント |
|---|---|---|
| 三重大学水産実験所 | 国立三重大学教育学部附属小学校 国立三重大学教育学部附属中学校 国立大学法人三重大学(医学部) 国立大学法人三重大学(教育学部) 国立大学法人三重大学(工学部) 国立大学法人三重大学(人文学部) 国立大学法人三重大学(生物資源学部) 三重大学教育学部附属養護学校 三重大学教育学部附属幼稚園 三重大学医学部附属病院 三重大学内郵便局 | 三重大学関連が対象になると思われるが、ぴったりの物は無い。LODAC側で新たに作る必要性あり。 |
| 北九州市立自然史博物館 | 北九州市立自然史・歴史博物館(いのちのたび博物館) | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 国立科学博物館 植物研究部 | 国立科学博物館 独立行政法人国立科学博物館附属自然教育園 国立科学博物館産業技術史資料情報センター 国立科学博物館分館 | たぶん、国立科学博物館をチョイスするのが妥当。 |
| 国立科学博物館(動物) | 国立科学博物館 独立行政法人国立科学博物館附属自然教育園 国立科学博物館産業技術史資料情報センター 国立科学博物館分館 | たぶん、国立科学博物館をチョイスするのが妥当。 |
| 北海道大学大学院水産科学研究院 | 北海道大学北方生物圏フィールド科学センター植物園 北海道大学総合博物館 北海道大学水産学部・水産資料館 北海道大学北方生物圏フィールド科学センター厚岸臨海実験所アイカップ自然史博物館 北海道大学(医学部) 北海道大学(教育学部) 北海道大学(経済学部) 北海道大学(工学部) 北海道大学(歯学部) 北海道大学(獣医学部) 北海道大学(水産学部) 北海道大学(農学部) 北海道大学(文学部) 北海道大学(法学部) 北海道大学(薬学部) 北海道大学(理学部) 北海道大学医学部附属病院 北海道大学歯学部附属病院 北海道大学前郵便局 | 北海道大学水産学部・水産資料館や北海道大学(水産学部)が近そうではあるが、ちょっと違う。やはり、LODAC側で新たに作る必要がありそう。 |
| 国立科学博物館(動物・人類) | 国立科学博物館 独立行政法人国立科学博物館附属自然教育園 国立科学博物館産業技術史資料情報センター 国立科学博物館分館 | たぶん、国立科学博物館をチョイスするのが妥当。 |
| 国立科学博物館(植物) | 国立科学博物館 独立行政法人国立科学博物館附属自然教育園 国立科学博物館産業技術史資料情報センター 国立科学博物館分館 | たぶん、国立科学博物館をチョイスするのが妥当。 |
| 筑波大学生命環境科学研究科 | 筑波大学附属図書館 国立筑波大学附属坂戸高等学校 国立筑波大学附属小学校 国立筑波大学附属中学校 国立筑波大学附属駒場中学校 国立筑波大学附属高等学校 国立筑波大学附属駒場高等学校 筑波大学附属盲学校 筑波大学附属大塚養護学校 筑波大学附属桐が丘養護学校 筑波大学(医学専門学群) 筑波大学(芸術専門学群) 筑波大学(図書館情報専門学群) 筑波大学(体育専門学群) 筑波大学(第一学群) 筑波大学(第三学群) 筑波大学(第二学群) 筑波大学附属病院 筑波大学内郵便局 筑波大学附属久里浜養護学校 筑波大学附属聾学校 | 筑波大学関連が対象になると思われるが、ぴったりの物は無い。LODAC側で新たに作る必要性あり。 |
| 静岡県自然学習資料保存事業室 | なし | LODAC側で新たに作る必要性あり。 |
| 茨城県自然博物館 | ミュージアムパーク茨城県自然博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 福井市自然博物館 | 福井市自然史博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 北九州市立自然史・歴史博物館 | 北九州市立自然史・歴史博物館(いのちのたび博物館) | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 名古屋大学博物館 | 国立名古屋大学教育学部附属中学校 国立名古屋大学教育学部附属高等学校 名古屋大学(教育学部) 名古屋大学(経済学部) 名古屋大学(工学部) 名古屋大学(情報文化学部) 名古屋大学(農学部) 名古屋大学(文学部) 名古屋大学(法学部) 名古屋大学(理学部) 名古屋大学(医学部) 名古屋大学医学部附属病院 名古屋大学内郵便局 名古屋大学病院内郵便局 名古屋大学 | 名古屋大学関連が対象になると思われるが、ぴったりの物は無い。LODAC側で新たに作る必要性あり。 |
| 多賀町立博物館 | 多賀町立博物館・多賀の自然と文化の館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 大阪市立自然史博物館(昆虫) | 大阪市立自然史博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 名古屋大学 | 名古屋大学 | LODAC側の情報として、検索条件としてfoaf:Organizationが無かったのがヒットしなかった原因。 |
| 大阪市立自然史博物館(動物・地学) | 大阪市立自然史博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 大阪市立自然史博物館(植物) | 大阪市立自然史博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 兵庫県立人と自然の博物館(植物) | 兵庫県立人と自然の博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 兵庫県立人と自然の博物館(その他) | 兵庫県立人と自然の博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 倉敷市立自然史博物館(以前はKMNH) | 倉敷市立自然史博物館 | 表記揺れがヒットしなかった原因。Silkのマッチングの閾値とかをいじれば、マッチングするかもしれない。 |
| 芸北 高原の自然館 | なし | LODAC側で新たに作る必要性あり。 |
| 九州大学 | 九州大学附属図書館 九州大学医療技術短期大学部 九州大学(医学部) 九州大学(教育学部) 九州大学(経済学部) 九州大学(工学部) 九州大学(歯学部) 九州大学(農学部) 九州大学(文学部) 九州大学(法学部) 九州大学(薬学部) 九州大学(理学部) 九州大学(芸術工学部) 九州大学医学部附属病院 九州大学歯学部附属病院 南九州大学(健康栄養学部) 南九州大学(園芸学部) 南九州大学(環境造園学部) 九州大学病院別府先進医療センター 西九州大学(健康福祉学部) | 九州大学関連が対象になると思われるが、ぴったりの物は無い。LODAC側で新たに作る必要性あり。 |
| 富山市科学博物館 | なし | LODAC側で新たに作る必要性あり。 |
| 静岡県自然学習資料センター | なし | LODAC側で新たに作る必要性あり。 |
- リンクの可能性
仮サイトで、Papilio xuthus(参考)で検索すると結果は30件で、ぴったり合致する物は無し。ナミアゲハで検索するとPapilioがヒット。この結果から和名を使ってマッチングするのが良さそうと判断できる。Silkを使って、完全合致の物、ある程度揺らぎを許容した物をリストし、チェックしてみようと思う。
TODO
- 元データの問題
scientific_termsのほうを見ていると謎な項目が結構あるがこれは 全部NIIのオンライン学術用語集由来.
@0034851@ is @異義語@ for @50%致死線量@ @生薬用語に対する英語の欠損@ is @生薬用語に対する英語の欠損@ for @アキョウ@ @R@ is @備考@ for @無触手綱@
番号はID.Rはそのまま記述されている. オンライン学術用語集の元データから日本語の異義語関係をIDベースに作成中.
- データのクリーニング
()が最初と最後についているついていない,記号のエスケープ(?)など
- ウェブサイト作成
http://lod.ac/bdls/ に仮作成した. まだ由来とかはまともにみられない.
- 外部リンク
CC-BY-SAだし,外部リンク作ればLOD cloud入れられるはずなので,リンク先を考える DBPedia他? Silkのいい実験になるかも.
メンバー
- 川本
- 南
- 加藤
(別プロジェクトデータ) 南極の苔データ
BH11.11/コケ 参照
写真
http://lod.ac/antmoss/images/xxxx
データ
http://togodb2.dbcls.jp/togodb/view/moss_test に試しに入れてみてある. entry数: 57000程度 RDF生成に1時間程度?