提供:TogoWiki
目的
- DBCLSにおいて環境などの様々なデータを取り込む上でRDFを利用しゲノム座標系に対してのデータ統合やアノテーション編集が可能なゲノムデータベースの開発試作
- 統合DB微生物グループにおいてデータ連携・統合に向けたRDF開発と改良
作業手順
- ゲノムDB毎のゲノム情報レポートを構成する要素(Stanza)の調査および整理
- UniProtとCyanoBaseから抽出したstanza確認 -11/21
- RDF表現のDB調査 -11/21,22
- Stanzaを構成する元情報のリストおよびRDF要素をKeynoteにまとめ
- サンプルRDF開発、作成および収集
- Stanzaを構成する情報収集するSPARQL設計
- Visualizationの設計
作業項目
ゲノムDB毎のゲノム情報レポートを構成する要素(Stanza)の調査および整理 -11/21
- UniProtとCyanoBaseから抽出したstanza確認 - 事前調査済 -ktym, so
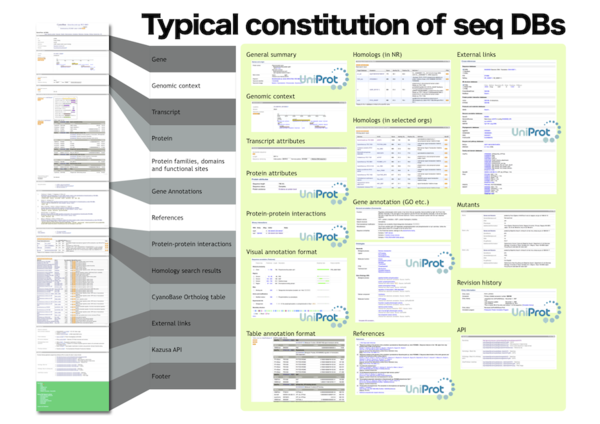
Typical constitution of seq DBs
- General summary of organisms
- General summary
- Genomic context
- Transcript attributes
- Protein attributes
- Protein-protein interactions
- Visual annotation format
- Table annotation format
- Homologs (in NR)
- Homologs (in selected orgs)
- Gene annotation (GO etc.)
- References
- External links
- Mutants
- Revision history
- API
RDF表現等のDB調査 -11/21,22
- その他の参考DB
- Refseq
- ensembl, UCSC -内藤さん siRNA、犬田 SRA
- WormBase -GFF3
- GFF3 Id/Parent関係表現しているRDF調査
セマンティックゲノムアノテーションデータベースで表現したいSOデータタイプの確認 -11/21,22
- replicon (SO:0001235)
- chromosome (SO:0000340)
- plasmid (SO:0000155)
- gene (SO:0000704)
- gene_member_region (SO:0000831)
- transcript (SO:0000673) -- CDS (SO:0000316) -- polypeptide (SO:0000104)
- ncRNA_gene (SO:0001263)
- tRNA (SO:0000253)
- rRNA (SO:0000252)
- small_regulatory_ncRNA (SO:0000370)
- protein_coding_gene (SO:0001217)
- pseudogene (SO:0000336)
- predicted_gene (SO:0000996)
- fusion_gene (SO:0000287)
- gene_with_polycistronic_transcript (SO:0000690)
- gene_with_trans_spliced_transcript (SO:0000459)
- mobile_genetic_element (SO:0001037)
参考
StanzaとGTPS (INSDC feature/qualifer) およびUniProtの要素との対応表 -11/22,23
Stanza対応表(書きかけ) [Todo]
Stanzaを構成するRDF設計 -11/21
- 全員:元となるゲノム・遺伝子のデータには GTPS や RefSeq が候補としてあげられた
- 全員:このうち GTPS をベースとすることが合意された
- 重元:GTPS の RDF に対し、feature 毎に ID を付与、適切な SO を付与、SO を利用した exon 単位での遺伝子モデル表現を追加
- 内山:GTPS と RefSeq の対応を location ベースで判定(3' の同一性を基準)、まずは syn についてサンプルデータを生成
- 川島:GTPS からは Protein ID がとれる。UniProt の RDF には embl-cds に Protein ID があるので、UniProt の RDF があれば対応可能
<rdf:Description rdf:about="http://purl.uniprot.org/uniprot/Q55168">
<rdfs:seeAlso rdf:resource="http://purl.uniprot.org/embl-cds/BAA10307.1"/>
- 渡辺:その他の UniProt から使えそうなデータについては BH11.11/セマンティックゲノムアノテーションデータベースの試作/UniProt にまとめる
- 片山:文献については UniProt のエントリにあるものはそれを活用すればよいし、TogoWS の PMID → Turtle 生成で RDF 化できる(subject の pmid:7890027 が QName として不正なのを修正する)
- 片山:Gene Ontology については、BH11.11/統合検索とRDFからの表データ生成#Gene_Ontology で geneontology.org の OWL, RDF のインポートを試みている(が現状では失敗)
- 岡本:微生物単位での環境アノテーションを用意
- 岡本:syn と ana について gene index による遺伝子 ID と文献 ID の対応を用意
- 内山:GTPS に含まれるゲノム間でのオーソログ対応のデータを用意
- 川島:OMIM については LinkDB に omim-kegg-hsa のデータがあるため、NCBI GeneID との対応は可能。OMIM の今後の更新は不明
これで少なくとも、微生物統合に必要なゲノムと遺伝子セットについて、UniProt + α の情報をもつ RDF ベースのデータベースを構築でき、環境などメタデータの付与や、遺伝子予測レベルごとのファセットブラウジング、統計的なデータの可視化、新規アノテーション付けとデータ公開までいけそう。
GTPS-Refseq対応表のRDF化 -11/22
TogoDB2 で変換して http://bh.dbcls.jp/ に投入した
select ?gtps ?refseq where {
?s <http://togodb.dbcls.jp/refseq_gtps_syn/metadata/gtps_gene> ?gtps .
?s <http://togodb.dbcls.jp/refseq_gtps_syn/metadata/refseq_gene> ?refseq .
}
| gtps | refseq
|
| ST15 | SLR0316
|
| ST12 | SLR0099
|
| ST2 | SLL0488
|
| : | :
|
GTPS の ID に生物種名を prefix しないと GTPS との RDF と対応が取れないということで
select ?gtps ?refseq
from <http://refseq/>
where {
?s <http://togodb.dbcls.jp/refseq_gtps_syn/metadata/gtps_gene> ?gtps .
?s <http://togodb.dbcls.jp/refseq_gtps_syn/metadata/refseq_gene> ?refseq .
}
| gtps | refseq
|
| Syne_PCC6803:ST1975 | SLR0954
|
| Syne_PCC6803:ST517 | SLL0993
|
| Syne_PCC6803:PL2919 | SLL0508
|
| : | :
|
データベースのID表記の整理 (Syechocystis sp. PCC 6803, slr1311)-11/24,11/25
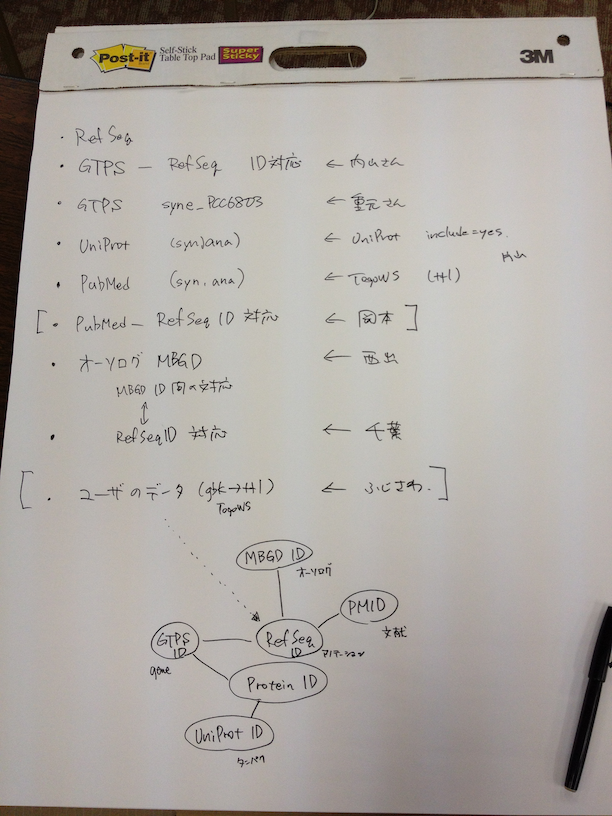
Synechocystis sp. PCC 6803を用いたサンプルデータの作成および収集 -11/21,22,23,24,25
| 型
| データ
| 担当者
| RDF化
| データストア
|
| ID関係
| GTPS-Refseq対応表
| 内山
| 済(togodb→turtle変換)
| 済
|
| ゲノム/遺伝子
| GTPS (syn)
| 重元
| 済
|
|
| ゲノム/遺伝子
| Refseq (syn)
| 片山
| 済
|
|
| タンパク質
| UniProt (syn.ana)
| UniProt
| 済
| 済
|
| ID関係
| Refseq ID-Pubmed対応表 (Gene Indexing、syn.ana)
| 岡本
| 済
| 済
|
| 文献
| Pubmed (syn.ana)
| TogoWS
| 済
| 済
|
| ID関係
| MBGD ID-Refseq ID対応表
| 千葉
| 済(togodb→turtle変換)
|
|
| オーソロググループ
| MBGD (syn)
| 西出
| 済
|
|
| 転写産物
| ユーザのデータ(例、HessさんのSyn6803TSS.gbk)
| 藤澤
| 済(togows/convert/genbank.ttl変換)
|
|
サンプルデータを用いた実装
Visualizationの設計
応用例
- 微生物統合DB(GTPS)に対して BLAST 検索した結果を RDF に集積されたデータを活用して enrichment
- ゲノムの領域を指定して領域に含まれるデータを返す API を用意し、BioDAS を超える。メタゲノムや RNA-Seq のマッピングデータを効率的にストリーミング。genoDiveで可視化
- 1つ以上のゲノムを1エントリーとしたゲノム差分情報を表現したデータベース開発 -11/25追加
作業ログ
11/23
- Sequence Ontology-GTPS Feature/qualifier対応作成と確認
- GTPS RDF表現の変更とサンプルデータ作成
rapper -o dot http://www.uniprot.org/uniprot/P16033.rdf?include=yes >P16033y.dot
dot -Tgif P16033y.dot -o P16033y.gif
11/24
- GTPS/RDF修正および議論
- INSDのCDSとの比較情報(grade)とBLASTやInterProScan、アノテーションによるフラグ情報(flag)の要素をばらして表現 ←ファセッティングでの利用を想定
- DDBJやGTPSのRDF表現で現在、ddbj名前空間をINSDCとする方向が望ましいが、現時点ではinsdc.orgにitemを指すURIが存在しないため変更なしとした
- MBGD/RDF開発
- Syn6803TSS.gbk/RDF作成
- togowsでgenbank形式ファイルをturtle形式ファイルに変換した
wget --post-file Syn6803TSS.gbk http://togows.dbcls.jp/convert/genbank.ttl
- 名前空間ns0はinsdcが望ましいのでは?という議論
<?xml version="1.0" encoding="utf-8"?>
<rdf:RDF xmlns:rdf="http://www.w3.org/1999/02/22-rdf-syntax-ns#">
<rdf:Description rdf:nodeID="g641410638">
<rdf:type rdf:resource="http://sabi.ddbj.nig.ac.jp/core/feature/TATA"/>
</rdf:Description>
<rdf:Description rdf:nodeID="g641410638">
<ns0:locus_tag xmlns:ns0="http://sabi.ddbj.nig.ac.jp/core/qualifier/">slr1179</ns0:locus_tag>
</rdf:Description>
<rdf:Description rdf:nodeID="g638353598">
<ns0:note xmlns:ns0="http://sabi.ddbj.nig.ac.jp/core/qualifier/">hypothetical protein </ns0:note>
</rdf:Description>
<rdf:Description rdf:nodeID="g638353598">
<ns0:product xmlns:ns0="http://sabi.ddbj.nig.ac.jp/core/qualifier/">hypothetical protein</ns0:product> </rdf:Description>
<rdf:Description rdf:nodeID="g638353598">
<ns0:db_xref xmlns:ns0="http://sabi.ddbj.nig.ac.jp/core/qualifier/">GeneID:953832</ns0:db_xref>
</rdf:Description>
<rdf:Description rdf:nodeID="g638353598">
<rdf:type rdf:resource="http://sabi.ddbj.nig.ac.jp/core/feature/CDS"/>
</rdf:Description>
<rdf:Description rdf:nodeID="g638353598">
<ns0:locus_tag xmlns:ns0="http://sabi.ddbj.nig.ac.jp/core/qualifier/">slr1394</ns0:locus_tag>
</rdf:Description>
<rdf:Description rdf:nodeID="g638353598">
<ns0:translation xmlns:ns0="http://sabi.ddbj.nig.ac.jp/core/qualifier/">MQDFGAVFHNQVLLISLAACFLAQGIKAIVEIFRNGKINLRSLVSTGGMPSAHSALVGALATGVGLQKGWGSNEFAIACLFAVIVMYDAAGVRQAAGKQARILNQLIDELFQEDQSLTEERLKELLGHTPVQVFAGLALGIAIAFFAVPAQ</ns0:translation>
</rdf:Description>
11/25
- Syn6803TSS.ttl作成
- MBGD ID-Refseq ID対応表
- MBGD/RDFのMBGD geneIDがURI表記のため対応表をURIに変更した
perl -ne 's/\t/,/;s/^/http:\/\/mbgd.genome.ad.jp\/rdf\/gene\//;print $_;' mbgdGeneId_refseqGeneId.syn.3625 > mbgdGeneId_refseqGeneId.syn.3625.csv
- togows/convert/csv.ttlを使ってみたらURLが変換されて新たなURLが生成された
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
<http://togodb.dbcls.jp/212974066c90b484cd52b4ec49679ab8/show/1>
<http://togodb.dbcls.jp/212974066c90b484cd52b4ec49679ab8/metadata/NP_942289.1> "NP_942157.1" ;
<http://togodb.dbcls.jp/212974066c90b484cd52b4ec49679ab8/metadata/http___mbgd.genome.ad.jp_rdf_gene_syn_SLL7001> "http://mbgd.genome.ad.jp/rdf/gene/syn:SSL5001" .
<http://togodb.dbcls.jp/212974066c90b484cd52b4ec49679ab8/show/10>
<http://togodb.dbcls.jp/212974066c90b484cd52b4ec49679ab8/metadata/NP_942289.1> "NP_942292.1" ;
<http://togodb.dbcls.jp/212974066c90b484cd52b4ec49679ab8/metadata/http___mbgd.genome.ad.jp_rdf_gene_syn_SLL7001> "http://mbgd.genome.ad.jp/rdf/gene/syn:SSL7004" .
- togodb2に入れてみた
- togodbでtutleの取得方法を片山さんに聞く
- コンフィグメニューのreleaseをクリックするとturtleが作成された
- 対応表のRefseq IDはRefseq proteinのAccessionのため千葉さんに確認
- mbgdにおいてrefseqのgene idおよびprotein idを確認して対応表を作り直した
- togodb2を利用してturtleファイルの作成した
@prefix rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#> .
<http://togodb.dbcls.jp/mbgd_refseq_syn/show/1>
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/mbgd> <http://mbgd.genome.ad.jp/rdf/gene/syn:SLL7001> ;
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/refseq_gene> "SLL7001" ;
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/refseq_protein> "NP_942289.1" .
<http://togodb.dbcls.jp/mbgd_refseq_syn/show/10>
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/mbgd> <http://mbgd.genome.ad.jp/rdf/gene/syn:SLR0613> ;
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/refseq_gene> "SLR0613" ;
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/refseq_protein> "NP_439901.1" .
<http://togodb.dbcls.jp/mbgd_refseq_syn/show/100>
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/mbgd> <http://mbgd.genome.ad.jp/rdf/gene/syn:SLR5016> ;
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/refseq_gene> "SLR5016" ;
<http://togodb.dbcls.jp/mbgd_refseq_syn/metadata/refseq_protein> "NP_942172.1" .
BH11.11での成果
- 統合DB微生物のデータ連携・統合に向けたRDF開発と改善の実施
- DDBJ/RDF形式をベースにGTPS/RDFの開発およびデータ統合および利用を想定し7項目についてRDFの表現の見直しおよび変更を実施した
- オーソログデータベースMBGDのRDF開発および3項目についての表現の変更を実施
- セマンティクゲノムアノテーションデータベース試作のためのSynechocystis sp. PCC 6803のRDFデータの作成および収集が完了した
課題
- データ統合する上で適切な名前空間、ID表現方法、rdf:typeに関するガイドライン策定
- stanza毎のSPARQL設計およびRDFのチューニング
- GFF3 Id/Parent関係表現についてのRDF開発
メンバ

