提供:TogoWiki
現状と今後の計画
- 副作用(臨床情報)から化合物経由で手持ちの遺伝子発現情報(ラットの実験による非臨床情報)への統合を行っています。
【超単純化したイメージ図】
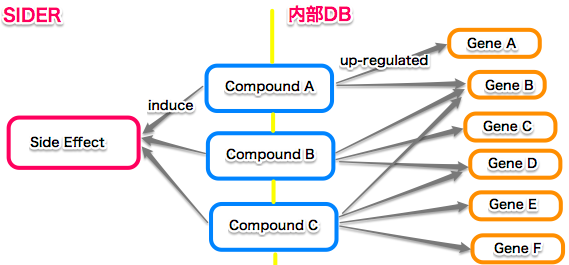
- 叩き台的なアプリケーション(副作用→上昇遺伝子 with その時投与された化合物 = 副作用を引き起こした化合物)を作成しました。
- 今後の方針として,得られた遺伝子に共通する特徴,機能等を得られるようなアプリケーションを作成しようと思っています。
相談
- マイクロアレイの実験条件に対して遺伝子発現情報(発現上昇/下降),遺伝子を付与する方法
- 現状,実験条件に対してpredicateにup-regulationを使い,直接遺伝子名を書いてしまっているのですが,修正が必要と感じています。
- プローブと遺伝子名,発現上昇/下降を実験条件に付与するための既存の例(あるいは役立つ例)はあるのでしょうか?
- 川島さんよりGene Expression Atlasを紹介して頂く。調査開始。=> "調査"へ
- 遺伝子に関連する情報を外部のDBからSPARQL 経由で得る方法
調査
PREFIX atlasterms: <http://rdf.ebi.ac.uk/terms/atlas/>
SELECT distinct ?analysis ?value ?v ?o
WHERE {
?analysis atlasterms:hasExpressionValue ?value .
?value ?v ?o.
}
LIMIT 100
PREFIX atlas: <http://rdf.ebi.ac.uk/resource/atlas/>
PREFIX atlasterms: <http://rdf.ebi.ac.uk/terms/atlas/>
SELECT distinct ?v ?o
WHERE {
atlas:A-AFFY-25:1390710_x_at ?v ?o.
}
LIMIT 100
- 一部の検索結果だけをピックアップしたところ,必要なスキーマは設計されている様子。
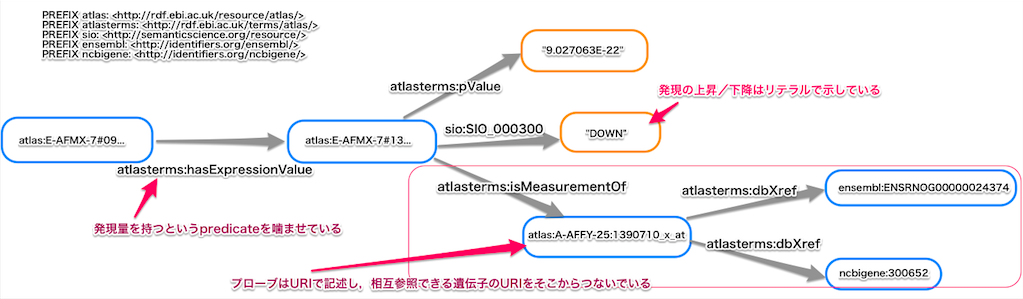
- ただ,Gene Expression Atlasは,今後もっと数値を使ったスキーマに設計され直されるのではないか,ということ(バイ川島さん)
進捗と今後の方針
- 現在のところ,数値を直接SPARQLでは使わないので,特定の条件で区切ってしまうことに。(例:log2foldの値 > 1 ,p-value < 0.05 で発現上昇と規定)
- GeneExpressionAtlasに習って,実験条件のRDFに対して,hasExpressionValueをpredicateとして付与し,p-value, Up, Down等も記述してしまう。
- 厳密に考えると実験条件の中の個体差の扱いもあるので,(同条件での実験でも個体差を考慮するためにラットを3匹使用していたりする)これらを同一に扱うか(平均値を取る)別々に扱うか否かは要相談&検討。

