BH11.11/ヒト・環境メタゲノムメタデータのオントロジー整備とRDF化
提供:TogoWiki
目次 |
目標
- ヒトメタゲノム・環境メタゲノムのメタデータの内容を整理し、既存の制御語彙もしくはオントロジーと対応付ける方法を考える
- 上記で設計した手法を実際のデータに適用しRDFデータをつくる
課題
- ヒトメタゲノム、環境メタゲノムのメタデータに記述されている内容(カテゴリー内容)について検討する -mri
- メタデータの内容の統計値
- ヒトメタゲノム 7万サンプル 85 カテゴリー
- 環境メタゲノム 4054サンプル 627 カテゴリー
- マニュアルキュレーションでオントロジーなどと関連付けると有用なカテゴリー項目の選定
- sanger (DDBJ FlatFile)の内容を検討 -kmn
- メタデータの内容の統計値
- オントロジーとの対応方法の検討 -so, yy, kwsm
- マッピング手法の調査検討とプロトタイピング -so, yy, kwsm
- NBRC菌株メタデータの内容と整理について -sgw
- 分離源、培地情報の整理とRDF設計
成果
- 関連研究、プロジェクト情報の調査
- 環境メタゲノム実験のメタデータの収集と整理 4054サンプル 627 カテゴリー
- 環境メタゲノム実験の生育場所情報の各種既存オントロジーへのマッピング 11434語→401語
- マッピング作業手順ワークフロー、マニュアルの整備
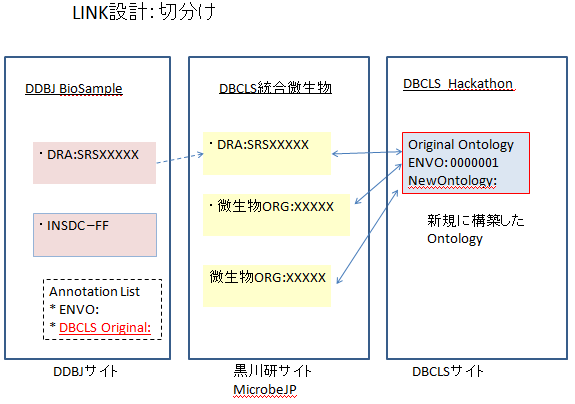
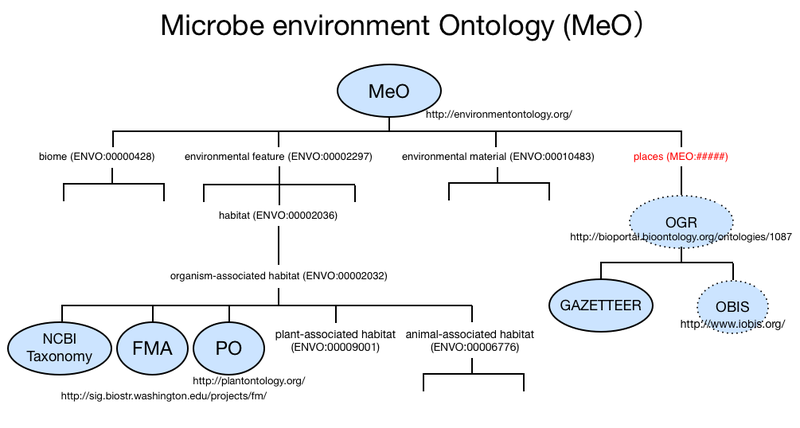
- 環境メタゲノム実験の生育場所を記述するオントロジーの設計 (MeO: Metagenome environmental Ontology)
- MeOによる環境メタゲノムへのアノテーション 1961サンプル(生育場所に関する情報のあるサンプル)
- 環境メタゲノム実験のメタデータOWL/RDF化 約8000サンプル emg_beta1.ttl一部サンプル
- 作成したRDFデータの利用方法の検討
- SPAQL エンドポイントの公開
Default Graph IRI: http://dbcls.jp/emg/
prefix meo: <http://dbcls.jp/meo/> prefix envo: <http://purl.obolibrary.org/obo/> prefix bs: <http://www.ncbi.nlm.nih.gov/biosample/> prefix emg: <http://dbcls.jp/env_metagenome/> prefix dbcore: <http://biodbcore.org/> prefix obo: <http://purl.obolibrary.org/obo/> SELECT ?s ?title ?place WHERE { ?s dbcore:id ?id . ?s dbcore:title ?title . ?s meo:environmental_feature ?habitat1 . ?s meo:environmental_feature ?habitat2 . ?habitat1 ?p "marine habitat"^^<http://www.w3.org/2001/XMLSchema#string> . ?s meo:place ?place . }
関連・支流
- BioProject, BioSample ヒアリング -kmn
- DDBJ(GenBank flatfile)のENV divisionデータの整理:
- ENV division = The ENV division of GenBank, for non-WGS sequences obtained via environmental sampling methods in which the source organism is unknown, debuted with release 147 in April 2005. (Benson et al. 2006) Sangerシークエンサーによるメタゲノム解析やメタ16S解析のデータなどが含まれる。
- DDBJ(GenBank flatfile)のENV divisionデータの整理:
Release 87.0(2011/9版) SOURCE /isolation_source, Accession数=約370万, ユニークタグ数=18,428
- 論文マイニングによる元データの拡張 -ota
- 論文出てるメタゲらしきデータ。57個。 http://g86.dbcls.jp/~inut/dono/publication_metagenome.html
- 「メタゲらしき」 means whose scientific name for given taxon id includes string "metagenome"
- study_type="metagenomics" なデータで、論文が出てるもの、ジャーナル一覧。
- proceedings of the national academy of sciences of the united states of america 13
- plos one 13
- environmental microbiology 6
- plos biology 5
- bmc genomics 5
- nature 4
- isme j 4
- bmc microbiology 4
- applied and environmental microbiology 4
- plos pathogens 3
- mol ecol 3
- j virol 3
- genome research 3
- science 2
- applied microbiology and biotechnology 2
- sci transl med 1
- plos neglected tropical diseases 1
- plos genet 1
- nucleic acids res 1
- nat med 1
- microb ecol 1
- j clin invest 1
- genome biology 1
- frontiers in microbiology 1
- fems microbiol ecol 1
- fems immunol med microbiology 1
- emerg infect dis 1
- bmc biology 1
- biodegradation 1
- astrobiology 1
- metagenomicsだと微生物だけでなくてウイルスメタゲ的なのも入ってしまうのと、WGS, RNA-seq等の別のstudy_typeに入ったやつは入らないのでたぶん網羅出来てはいません。上の一覧はあくまでどの辺りへの投稿が多めなのか調査です。
- PubmedCentralで全文がフリーで公開されているのでとりあえずPMCと、以前やりかけていたPLoS, BMC系ジャーナルのHTMLパーサー書いた。
- じゃあ三本でmetagenomics絡み論文をばとりあえず斬ってみませうかというあたりでタイムアップ…
- SRAデータ付随のメタデータよりも多く、正確な情報が追加されることを期待していますが、実際どのくらい補えるかはわかりません…
- これまで見てきた感じだと、論文が出てるデータはsubmissionもしっかりしていてメタデータ親切に書いてくれてるケースが多いような気がします[要出展]
- supplementaryのExcel, Word, and pdfなどのダウンロードして見る系のデータをplain textにconvertするようにもしました
- figure legendやテーブルの中の文字までプレーンに起こせば機械的にexact matchのオントロジに当てたり出来ないかな、どうかな的。
- 切ったものをファイルとして吐くところ or web interfaceがあとちょっと残ってるので、それが終わったらここにGitHubなりで諸々置いておきます。
- 論文出てるメタゲらしきデータ。57個。 http://g86.dbcls.jp/~inut/dono/publication_metagenome.html
将来的な連携?
- メタデータによる各機関のリソース連携案 -kmn
作業ログ
2011-11-21
- メタデータのどのカテゴリーを対象とするか
- 環境メタゲノムの生育場所に関連する情報を対象にする
- どのようなオントロジーを使うか
- 先行研究のサーベイ
- Genome Standard ConsortiumでのEnvOとGOLDのマッピング試み
- GOLDにおけるゲノム・メタゲノムのHabitatデータとEnvOとのマッピング 方向性は同じ、違いはRDF化するかしないか、predicateに何を使うか、など。
- PhenomeNet Robert論文
- 5255株の細菌について、分離源をEnvO-Liteで表現した試み 分離源の記述にもEnvO-Liteが用いられている。
- EnvO-Lite 20タームのみ定義されている。Habitatの大分類としては使えるかも。
- MG-RASTのメタゲノムメタデータ検索 メタデータをXML形式で記述し、project_descriptionの文章を全文検索してユーザが入力したワードにマッチするメタゲノムサンプルを一覧表示。オントロジーは特に使っていない。環境の大まかな分類(Biome)は、EnvO-Liteを用いているように見える。
- カテゴリー選抜した値のみをBioPortal Ontolo gy Recommenderに再度投げて推薦オントロジーの内容を精査する
- 検索結果のオントロジー規模の偏りを標準化する秘密のコマンドを金さんに聞くこと
- 先行研究のサーベイ
- マッピング作業の設計
- 作業設計のまとめ
- 利用ボキャブラリー, オントロジー, predicate, を元に作業マニュアルを共有
- 作業分担
- 想定しているユースケースの列挙
- LODチャレンジへの応募を検討する
2011-11-22
- 森さんが作成した、1961 のSRSサンプルに対して、環境に関係するメタデータが記述された39カテゴリーのメタデータ一覧をEnvOにマッピング
- とりあえず、5人で、それぞれ30 SRSサンプルずつ、EnvOにマッピングし、それぞれの作業結果を全員でレビューした
- 1,961のサンプルに記述された環境メタデータの総数は3000程度
2011-11-23
- 森さんが、3000強あった、SRAの環境に関係しそうなタームを、400程度にまで減らしたリストを作成
- 400のタームについては、マニュアルでマッピングすることにした。
- 昨日、作業した120については、各自の担当分を記入するだけなので、残りは約300。=>ほぼ完成
- マップされたEnvO IDが、EnvOのどの階層に登場するか調査した.
- [/mw/index.php/BH11.11/ヒト・環境メタゲノムメタデータのオントロジー整備とRDF化/envo_hierarchy#EnvO.E3.81.AE.E9.9A.8E.E5.B1.A4.E6.A7.8B.E9.80.A0 EnvOの階層構造(第三階層まで)
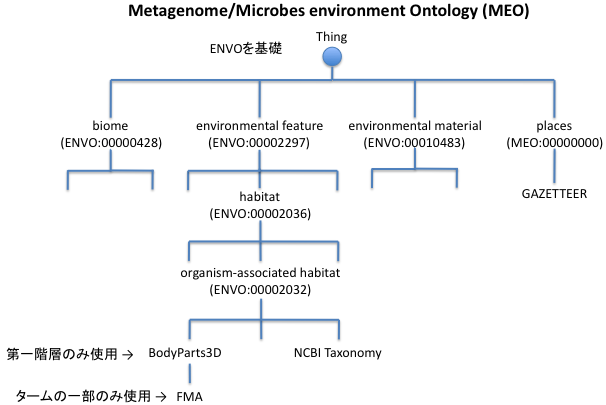
- 第一階層としては、"biome" (ENVO:00000428), "environmental feature" (ENVO:00002297), "environmental material" (ENVO:00010483) の3つだけであった。
- これらの結果から、メタゲノム・ゲノムの生育環境オントロジーをどのように設計するか考察した。
- オントロジー構造の変更
- 現状のEnvOだと足りない点を別のオントロジーで補う
- 候補としては、 場所のオントロジーとして、OGR: Ontology of Geographical Region や OBIS: Ocean Biogeographic Information System等。 Habitatの、アナトミー関連のオントロジーとして、FMA: Foundational Model of Anatomy ontology, PO: Plant Ontology 等。
- 参考リンク Wikipedia:Saline water
2011-11-24
- Microbe environment Ontology (MeO)のデザインを修正
- 追加オントロジーのマッピング
- マッピング作業の整理とマニュアル化
- マッピング作業支援ツールの開発
- ターム生成のためのフィルタ群 -yy
- オントロジーサーチ -kim
- LODチャレンジへの参加を検討
- TODO
- MeOのデザインの改善
- Protegeを用いてMeOをOW/RDF化する
- SRS アノテーション作業の設計
2011-11-25
- MeO作成のためのオントロジーマージ操作とOWL/RDF化 → Protege/BioPortalがうまく動作せず断念
- マッピングファイルの修正
- SRS IDに対するMeOを用いたアノテーションの設計
- SRS RDFの作成
- Virtuosoへのアップロードテスト
- まとめ
2012-1-31 (BH11.11終了後)
- マッピングファイルの修正
- MeO作成のためのオントロジーマージ操作とOWL化
- GAZETTEERのOBOtoOWL変換
- TopBraidComposerでのマージ作業に挑戦し失敗
- OBO-Edit2を用いてoboファイルをマージ+owlファイルへの変換
- SRS RDFの作成
- プロジェクトページの作成
- LODチャレンジへのSubmit
メンバー (所属) 興味
- 菅原先生 (DDBJ) 培地・分離源情報のRDF化 [sgw]
- 重元さん (DDBJ) GTPS, 各種RDF化
- 大田さん (DBCLS) SRAメタデータ整理 NGSデータ整理 [ota]
- 内藤さん (DBCLS) "GGRNA"高速配列検索, RNA配列, DNA配列
- 金さん(DBCLS)オントロジー, テキストマイニング [kim]
- 岡本 (DBCLS) アノテーション, キュレーション, 微生物ゲノム [so]
- 内山先生 (NIBB) オーソログ分類→メタゲノム, MBGD
- 西出さん (NIBB) MBGD開発実務
- 千葉さん (NIBB) MBGD開発実務, 微生物統合プロジェクト
- 川島さん (HGC) ゲノム環境オントロジー, LinkDB RDF化 [kwsm]
- 山本さん (DBCLS) Text Mining, 表記ゆれの吸収など [yy]
- 中村教授 (DDBJ) DRA, INSDC, 微生物ゲノム [yn]
- 神沼さん (DDBJ) DRA, DTA, DOR/CIBEX, BioSample, BioProject [kmn]
- 黒川先生(東工大)メタゲノム比較, microbe.jp, 微生物統合プロジェクト
- 森さん (東工大) メタゲノム比較, microbe.jp, 微生物統合プロジェクト [mri]
- 竹原さん (東工大) メタゲノム比較, 微生物統合プロジェクト
- 吉野さん (東工大) メタゲノム比較, 微生物統合プロジェクト
- 後藤さん (阪大) 病原菌メタゲノム, OpenBio