BH14.14/TogoGenome
提供:TogoWiki
- TogoGenome 真核対応
目次 |
stanza
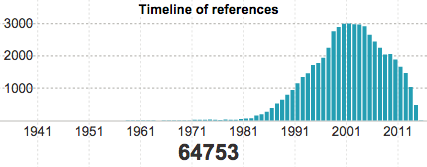
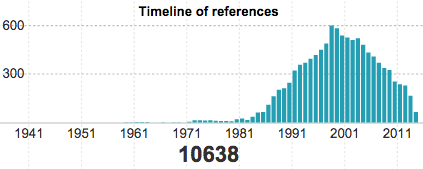
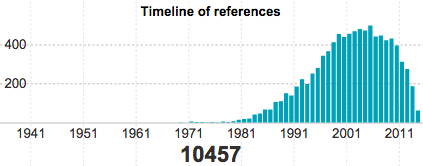
- 生物種単位の引用数のタイムライン nano stanza 作成
- 遺伝子リスト stanza に Reactome の情報追加
- 引用数がリストとタイムラインとで合わないバグ
- date を複数持つ citation があった(例 Epub 2010, publish 2011)
- 引用数がリストとタイムラインとで合わないバグ
LODEStarかvirtuosoのバグっぽい
- Reactome
- 指定階層以下の階層を取ってくる
- FILTER だと結果が得られない。type 指定だと取れる
- 行数が多いと filter のほうが早い
PREFIX bpx: <http://www.biopax.org/release/biopax-level3.owl#>
SELECT DISTINCT ?parent ?reactome
FROM <http://rdf.ebi.ac.uk/dataset/reactome/49>
WHERE {
?parent a bpx:Pathway .
FILTER CONTAINS(STR(?parent), "REACT_111217")
?parent bpx:pathwayComponent+ ?reactome .
FILTER CONTAINS(STR(?reactome),"REACT") # -> ?reactome a bpx:Pathway .
}
ORDER BY ?reactome
- DBCLSミラーだと一応どっちでも取れるけど、結果が異なるので virtoso にバグがある(グラフは古いので 49 -> r45)
- 下記のようにFILTER 一個だけだと error が出るので、virtuoso のバグに対する LODEStar の対応が不十分なだけかも
PREFIX bpx: <http://www.biopax.org/release/biopax-level3.owl#>
SELECT ?reactome
FROM <http://rdf.ebi.ac.uk/dataset/reactome/49>
WHERE {
<http://identifiers.org/reactome/REACT_111217.3> bpx:pathwayComponent+ ?reactome .
FILTER (REGEX(?reactome, "REACT"))
# ?reactome a bpx:Pathway .
}
ORDER BY ?reactome
- 結論
- virtuoso(7.10)でプラスとかアスタリスクで全部取ってきたものをフィルタリングすると、一部だけ取れてこないなど、気づかない感じで間違った結果になるようです
- LODEStar は危険なクエリですよと error を返してくれるけど、ときどきスルーして間違った結果になるようです
真核対応版 JBrowse 用 SPARQL
DEFINE sql:select-option "order"
PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#>
PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#>
PREFIX xsd: <http://www.w3.org/2001/XMLSchema#>
PREFIX obo: <http://purl.obolibrary.org/obo/>
PREFIX faldo: <http://biohackathon.org/resource/faldo#>
PREFIX insdc: <http://ddbj.nig.ac.jp/ontologies/sequence#>
SELECT DISTINCT ?start ?end ?strand ?type ?name ?description ?uniqueID ?parentUniqueID
FROM <http://togogenome.org/graph/refseq/>
FROM <http://togogenome.org/graph/so/>
FROM <http://togogenome.org/graph/faldo/>
WHERE
{
{
SELECT ?start ?end ?strand ?type ?uniqueID ?parentUniqueID
WHERE
{
?seq_id ?p "{ref}" .
?uniqueID obo:so_part_of+ ?seq_id .
FILTER ( !(?start > {end} || ?end < {start}) )
?uniqueID faldo:location ?loc .
?loc faldo:begin/faldo:position ?start .
?loc faldo:end/faldo:position ?end .
?loc faldo:begin/rdf:type ?faldo_type FILTER ( ?faldo_type IN (faldo:ForwardStrandPosition, faldo:ReverseStrandPosition, faldo:BothStrandsPosition) ).
BIND ( if(?faldo_type = faldo:ForwardStrandPosition, 1, if(?faldo_type = faldo:ReverseStrandPosition, -1, 0)) as ?strand )
?uniqueID rdf:type ?uniqueID_type FILTER ( ?uniqueID_type %SO% ).
?uniqueID_type rdfs:label ?uniqueID_type_label .
BIND ( str(?uniqueID_type_label) as ?type ) .
?uniqueID obo:so_part_of ?parentUniqueID %FILTER% .
}
}
OPTIONAL { ?uniqueID insdc:locus_tag ?name . }
OPTIONAL { ?uniqueID insdc:product ?description . }
}
を
DEFINE sql:select-option "order"
PREFIX rdf: <http://www.w3.org/1999/02/22-rdf-syntax-ns#>
PREFIX rdfs: <http://www.w3.org/2000/01/rdf-schema#>
PREFIX xsd: <http://www.w3.org/2001/XMLSchema#>
PREFIX obo: <http://purl.obolibrary.org/obo/>
PREFIX faldo: <http://biohackathon.org/resource/faldo#>
PREFIX insdc: <http://ddbj.nig.ac.jp/ontologies/nucleotide/>
SELECT DISTINCT ?start ?end ?strand ?type ?name ?description ?uniqueID ?parentUniqueID
FROM <http://togogenome.org/graph/refseq>
FROM <http://togogenome.org/graph/so>
FROM <http://togogenome.org/graph/faldo>
WHERE
{
?refseq_id insdc:sequence_version "{ref}" .
?refseq_id insdc:sequence ?seq_id .
?parentUniqueID obo:so_part_of* ?seq_id FILTER ( ?parentUniqueID != ?seq_id ) .
?parentUniqueID rdfs:subClassOf ?parentUniqueID_type FILTER ( ?parentUniqueID_type IN( obo:SO_0000316 ) ).
?parentUniqueID obo:so_has_part ?uniqueID .
?parentUniqueID_type rdfs:label ?parentUniqueID_type_label .
BIND ( str(?parentUniqueID_type_label) as ?type ) .
?uniqueID rdfs:subClassOf ?uniqueID_type FILTER ( ?uniqueID_type IN( obo:SO_0000147 ) ).
?uniqueID faldo:location ?loc .
?loc faldo:begin/faldo:position ?pos_start .
?loc faldo:end/faldo:position ?pos_end .
?loc faldo:begin/rdf:type ?faldo_type FILTER ( ?faldo_type IN (faldo:ForwardStrandPosition, faldo:ReverseStrandPosition, faldo:BothStrandsPosition) ).
BIND ( IF (?faldo_type = faldo:ForwardStrandPosition, 1, if(?faldo_type= faldo:ReverseStrandPosition, -1, 0)) as ?strand ) .
BIND ( IF (?faldo_type = faldo:ReverseStrandPosition, ?pos_end, ?pos_start ) AS ?start ).
BIND ( IF (!(?faldo_type = faldo:ReverseStrandPosition), ?pos_end , ?pos_start) AS ?end ).
FILTER ( !(?start > {end} || ?end < {start}) )
OPTIONAL { ?parentUniqueID rdfs:label ?name . }
OPTIONAL { ?parentUniqueID insdc:product ?description . }
}
として、真核に多い multiple exon (join のある feature) の遺伝子は拾えるようになったが、 single exon (join のない feature) については特別扱いが必要となることがわかったので、 insdc2ttl.rb を変更して single exon についても multiple exon と同様のトリプルを出力するようにし、 ↑の SPARQL だけでどちらにも対応できるようにすることになった。
また insdc2ttl.rb で /locus_tag, /gene から取得した遺伝子の ID を rdfs:label としていたが、 これにくわえ dc:identifier もつけることになった(川島さんの RDF 化ガイドラインにも沿う形)。
https://github.com/dbcls/rdfsummit/commit/f318bdb3baa4aac83f6351b3db788776c17b53a8
INSDC (RefSeq) to Turtle 更新
Gene が CDS (protein_coding) か tRNA (tRNA_coding) か ncRNA (non_protein_coding) かといった型をきちんと付けるように改良。さらに ncRNA の場合は qualifier の /ncRNA_class を見て、gRNA_encoding, miRNA_encoding, SRP_RNA_encoding などを利用するようにした。 (2015/2/6)
- stRNA_encoding については stRNA が ISNDC の feature にも qualifier にも出ないので保留
- snoRNA, snRNA, scRNA は現在 feature にあるが、いずれ ncRNA の qualifier に移動されるのではないかとのこと