BH13.13/TogoDB
提供:TogoWiki
目次 |
統合DB実務者会議
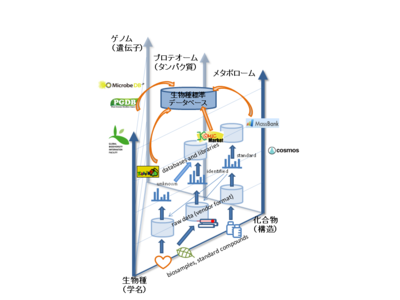
6グループ(金谷、黒川、成松、田畑、中村、豊田)の実務者で次期統合の横串を考えます。 メタボローム等の omics データを管理する時に必要な情報の共通化をはかります。
- 問題点
- 生物種の名称問題 ... NCBI Taxonomy で足りない部分
- 化合物の名称問題 ... InChI, CAS番号で足りない部分
参加グループのまとめ
金谷G
MassBank, BioMassBank, KNApSAcK の統合化。 代謝物の CAS ID は整理済みで、InChI コードも生成可能。
生物種を中心に情報収集してきたが、生物間相互作用の情報、生理活性情報も文献から収集。 生理活性情報に関しては既に RDF 化。
申請タイトル「生物種標準メタボローム・データベースの構築」
黒川G
MicrobeDB.jp としてオントロジーの統合化
- Gene
- MBGD, GTPS/RefSeq, TogoAnnotation
- Taxon
- NCBI Taxonomy, NBRC/JCM
- Environment
- INSDC SRA (metadata & metagenome)
配列相同性をもとに情報を統合、RDF化。菌株データは既に真核微生物(藻類、カビ等)も含まれているので、バクテリアのみならず真核微生物も含めることになりそう。 文献からの配列情報等を集めたら、あとは配列相同性で整理するのが楽かも。
成松G
糖鎖構造を統一的に記述する wurcs フォーマットがほぼ完成。GenBankのような糖鎖バンクを作りたい。 wurcs はアグリコンを自動的に取り除き、糖鎖部分が同じものをコード化してくれる。 植物二次代謝物の配糖体にも使えるかもしれない。
中村G
PDB は全て RDF 済み。さらに chemical component というセクションがあり、PDB に出てくる化合物毎に整理。InChI 完備。 糖鎖はタンパク質の精製条件等にも依存。
生物種の情報はあまり入っていないが、source 生物種に関しては NCBI Taxonomy 情報有り。
豊田→桝屋G
生物種の垣根を超えたフェノーム汎用スキームの構築 「何の(器官):性質が(形質):どうなった」
微生物が分子を産生するという情報も、表現型の1つとみなせる(分子表現型)。生理活性もある意味、表現型である。 フェノタイプという側面から連携したい。
田畑G
植物の遺伝子マーカー、オーソログ情報を地道にデータベース化。各データの規模はそう大きくない。 関連データベースの情報を取り込んでリンク、現在数多くのDBと連携。
次期は多くのデータへ統合化。全体を俯瞰するビジュアルの設計が重要。
統合の方向性
- 理想
- RDF による世界の救済
- 現実
- 集まってもらったグループの中だけでもRDF化で連携。全グループに共通するデータは、学名(生物種)と化合物名
生物種
- 実施事項
BioSample ID
- DDBJが企画する BioSample ID
- BioSample ID は原則、各 contributor まかせ(整理しきれない)
- 統合DBの枠組み内ではマウス、植物、微生物で分けて対応する必要
- 合意事項
遺伝子配列がある場合は NCBI Taxonomy があるので、このIDを共通化の際に用いる。 URIを揃えるため [1] を使うこと。
遺伝子配列がないものについては、なるべく新しい学名を用いる。といっても、論文における名前は信用できないので探す。
- 動物なら GBIF http://www.gbif.org
- 植物なら APG http://tolweb.org/Angiosperms/20646
- 微生物は LPLN http://www.bacterio.net/index.html
代謝物
- 実施事項
- 糖鎖に注目(糖脂質、糖タンパク質、二次代謝物)、糖転移酵素の分類と同定
- フラボノイドの構造 (7000件) に付随する糖鎖構造を wurcs フォーマットに変換
- 統合DBの時期申請に、揮発性化合物と昆虫の相互作用を明示的に含める(関連研究者)
- 合意事項
- CAS番号を少なくとも一つつけること
- InChI はMOLファイル(構造ファイル)依存なので、ChemSpider や PubChem で見つかる場合はそれをコピペすること
- 日化辞 ID もつけると日本語化が進んで良い
逆に言えば、化合物名だけは避ける。PubChem ID や ChemSpider ID だけに限定するのは避ける。